MALDI mass spectrometry imaging and in situ microproteomics of Listeria monocytogenes biofilms
Santos T1, Théron L2, Chambon C2, Viala D2, Centeno D2, Esbelin J1, Hébraud M3
1 Université Clermont Auvergne, INRA, UMR MEDiS, F-63122 Saint-Genès Champanelle, France
2 INRA, Plateforme d’Exploration du Métabolisme, composante protéomique (PFEMcp), F-63122 Saint-Genès Champanelle, France
3 Université Clermont Auvergne, INRA, UMR MEDiS, F-63122 Saint-Genès Champanelle, France; INRA, Plateforme d’Exploration du Métabolisme, composante protéomique (PFEMcp), F-63122 Saint-Genès Champanelle, France
Les protéines de surface constituent des cartes d’identité des cellules bactériennes et ont une importance particulière dans leur interaction avec l’environnement. Cependant, elles sont très difficiles à caractériser et à étudier par des techniques biochimiques classiques. Afin de lever ce verrou, une approche d’imagerie par spectrométrie de masse (MSI), généralement utilisée sur des tissus biologiques, a été expérimenté sur des biofilms de Listeria monocytogenes.
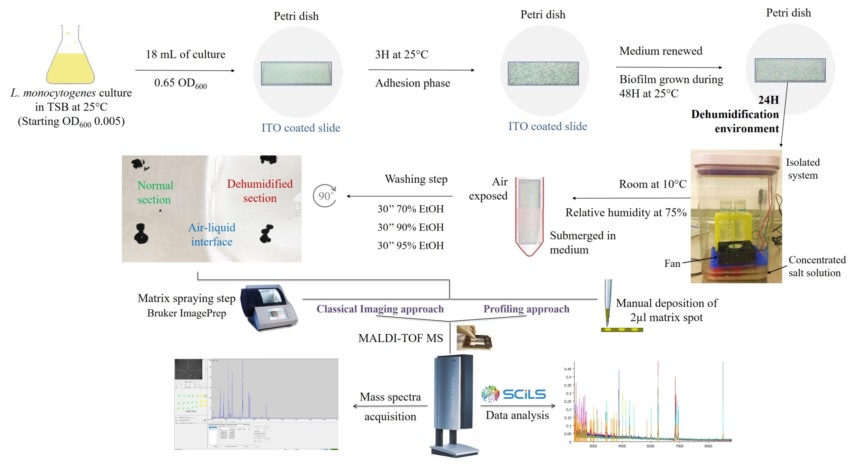
L’imagerie par spectrométrie de masse (MSI) est une technologie de cartographie de surface qui permet de déterminer l’information spatiale et l’abondance relative des analytes (protéines, peptides, lipides ou petits métabolites) directement à partir de la surface d’une coupe d’échantillon biologique. Initialement développée pour comparer des cartes ioniques et identifier des biomarqueurs de pathologies au sein des tissus biologiques à des fins médicales, la MSI est maintenant mis en œuvre dans de nombreux autres domaines. L’objectif de ce travail était de développer et d’optimiser un workflow (Figure) permettant l’utilisation de la MALDI MSI sur des biofilms bactériens puis, comme preuve de concept, de l’appliquer à l’étude de l’impact au niveau moléculaire d’un stress environnemental. En raison de la particularité du matériel biologique et des contraintes techniques associées, la mise en œuvre de cette technologie représentait un véritable défi auquel très peu de laboratoires se sont confrontés jusqu’à présent. Le stress appliqué consistait à imiter les conditions d’un atelier agroalimentaire sur des biofilms du pathogène Listeria monocytogenes, dans lequel l’air est déshumidifié après les opérations de nettoyage désinfection des surfaces et des équipements, afin d’éliminer l’eau résiduelle, d’assainir l’atmosphère ambiante et de limiter la propagation de la contamination.
Le protocole de MALDI MSI que nous avons développé a permis de comparer des sections de biofilms immergées dans un milieu de culture et des sections de biofilms déshumidifiées, maintenues dans une humidité relative de l’air de 75%. L’analyse a révélé la localisation spatiale de 47 protéines entre les deux sections, dans une gamme de masse allant de 2 à 15 kDa. Cependant, l’identification de la nature de ces protéines directement à partir des pics de MSI est très difficile. De ce fait, une approche de type « shotgun proteomics (LC-MS/MS) », découplée de la MSI, a été mise en œuvre permettant d’extraire plus de 500 protéines à la surface de l’échantillon biologique et de les identifier. Le recoupement des données de masse obtenues par les deux techniques (« back-correlation ») a ensuite conduit à valider et à identifier la nature de 5 des 47 protéines révélées par MSI.
Ce travail démontre comment la MALDI MSI peut être utilisée pour disséquer le protéome d’un biofilm bactérien intact, donnant ainsi de nouvelles perspectives sur l’expression des protéines. D’un point de vue plus général, cette approche enrichit de façon prometteuse les différentes techniques et méthodologies déjà utilisées pour explorer et comprendre la physiologie des microorganismes dans les biofilms. Des développements méthodologiques seront poursuivis pour analyser les peptides résultant de l’hydrolyse trypsique des protéines à la surface des cellules bactériennes, dans le but d’améliorer leur identification.
Santos T, Théron L, Chambon C, Viala D, Centeno D, Esbelin J, Hébraud M. MALDI mass spectrometry imaging and in situ microproteomics of Listeria monocytogenes biofilms. J Proteomics. 2018 Sep 15;187:152-160. PMID : 30071319.