LE MICROBIOTE PULMONAIRE, UN ENJEU RECENT EN MICROBIOLOGIE MEDICALE
Un jeune microbiote avec une déjà longue histoire
L’histoire retiendra qu’il fut plus évident de prouver la présence de bactéries dans les abysses océaniques que dans les poumons humains sains. Casser un dogme centenaire (« un poumon sain est stérile ») n’est pas simple. Rétrospectivement, il est intéressant de s’interroger sur la construction de ce mythe ; l’histoire de la médecine est jonchée de détours, de pas en arrière et de bonds en avant bien souvent propulsés par des avancées technologiques. S’agissant du microbiote pulmonaire, force est de constater que, dès la fin du XIXème siècle, les données montrant une « flore pulmonaire » étaient disponibles chez l’Homme ou les mammifères ; mais les esprits n’étaient pas prêts. Ainsi, en 1899, l’étude bactériologique des poumons de 23 cadavres indemnes de toute affection pulmonaire avait identifié dans deux tiers des cas des bactéries (dont des streptocoques autres que le pneumocoque), soulignant comme « fait important et incontestable, la présence dans la plupart des poumons sains de microbes pathogènes habituels des voies respiratoires excluant leur pénétration agonique » (Beco 1899) ; la seule erreur dans cette conclusion prémonitoire fut le mot « pathogène » (Figure 1). Par la suite, bien d’autres études ont montré la présence de bactéries dans les prélèvements respiratoires de sujets sains ; il est probable que la découverte du rôle important de la clairance muco-ciliaire et des macrophages alvéolaires dans la clairance bactérienne décrit comme gardant les poumons « propres et stériles » (Green, 1964) soit venue brouiller l’analyse de ces données interprétées comme des contaminations par la sphère oropharyngée et non véritablement comme la preuve d’un microbiote pulmonaire résident. Il est vrai que depuis Pasteur et les premières heures de la microbiologie clinique, les analyses des prélèvements broncho-pulmonaires étaient surtout réalisées dans un contexte infectieux aigu (PFLA[1], grippe, etc.) ou chronique (mucoviscidose, BPCO[2], asthme, etc.). Preuve supplémentaire que l’idée même d’un microbiote pulmonaire sain n’était pas acceptée encore récemment, le premier « Human Microbiome Project », grand projet de description des microbiotes humains sains initié aux Etats-Unis par le NIH[3] en 2008, omit totalement le poumon. Il fallut attendre l’application des analyses métagénomiques (qui avaient déjà fait la preuve de leur puissance sur des prélèvements fécaux) aux pathologies respiratoires chroniques pour admettre que le signal microbien détecté dans les voies aériennes inférieures de sujets pris comme témoins n’était pas une contamination ou une infection, mais correspondait bel et bien à un microbiote pulmonaire. En 2010, une étude métagénomique de patients asthmatiques fit le constat, après analyse de LBA[4] des sujets du bras contrôle, de la présence de communautés bactériennes (Hilty 2010). Depuis, bien que peu nombreuses (du fait de la difficulté d’accès à l’organe), les études réalisées en situation physiologique s’accordent toutes sur l’existence d’un microbiote pulmonaire commensal (Basis 2013, Segal 2013, Dickson 2105). Ainsi, la vision binaire stérilité/pathogénicité a peu à peu cédé la place à celle d’un microbiote pulmonaire qui jouerait un rôle dans la physiologie pulmonaire.<
Mais qu’en savons-nous vraiment ? Quels micro-organismes composent ce microbiote ? Quels rôles remplissent-ils ? Représentent-ils de nouvelles opportunités en médecine ? Le microbiote pulmonaire commence à peine à livrer ses secrets.
Un microbiote de maturation précoce
Le débat sur le microbiote placentaire en dehors de toute situation obstétricale à risque infectieux n’est pas tranché et l’existence d’un microbiote pulmonaire in utero reste hypothétique bien qu’une étude récente réalisée chez 18 fœtus morts prématurément ait révélé la présence d’un microbiote pulmonaire dès le 1er trimestre de grossesse (Al Alam 2020). Une donnée est certaine : la colonisation des voies respiratoires commence a minima à la naissance. Des communautés microbiennes ont été détectées dans la cavité orale et le nasopharynx des nouveau-nés dans les 5 minutes qui suivent la naissance et dans les poumons dès 24 heures de vie (Dominguez-Bello 2010, Pattaroni 2018). La mise en place du microbiote pulmonaire est très rapide comparée à celle du microbiote intestinal. On a coutume de dire que tout se joue avant l’âge de 2 à 3 ans pour le microbiote intestinal ; pour le microbiote pulmonaire, c’est en mois qu’il faut compter. En à peine 2 mois de vie, le microbiote pulmonaire a achevé sa maturation (Pattaroni 2018). Dès les trois premiers jours de vie, trois profils de microbiote sont distinguables : un pulmotype diversifié et stable composé de Neisseria, Streptococcus, Prevotella, Veillonella, Fusobacterium et Porphyromonas et associé à une naissance à terme, un second caractérisé par Ureaplasma chez les enfants nés par voie basse et un troisième caractérisé par Staphylococcus chez les enfants nés par césarienne ; ces deux derniers pulmotypes étant temporaires et associés à la prématurité. Chez les grands prématurés souffrant de bronchodysplasie, on observe à l’âge adulte une dysbiose pulmonaire avec une nette diminution de l’abondance relative de Prevotella (Rofael 2019). Ces données suggèrent que l’âge gestationnel, le mode d’accouchement et les deux premiers mois de vie sont cruciaux dans la constitution du microbiote pulmonaire de l’adulte.
Un microbiote unique par ses caractéristiques
La niche microbienne pulmonaire est complexe ne serait-ce que par l’anatomie de l’organe et la surface que représente le tractus respiratoire (60 à 80 m2). Il est important de souligner qu’à la diversité anatomique (trachée, bronches, bronchioles, sacs alvéolaires, etc.) correspond une véritable biogéographie dont la complexité a été difficile à appréhender à ce jour, en dehors d’études réalisées sur explants respiratoires ou sur des prélèvements étagés (Charlson 2011, 2012, Dickson 2017). Les sources microbiennes qui alimentent cette niche sont multiples : le réservoir bucco-dentaire en premier lieu, le réservoir digestif et l’air inhalé. Le microbiote pulmonaire est la résultante d’une balance entre immigration (par inhalation, micro-aspiration, dispersion à partir de la muqueuse oropharyngée) et élimination (par l’ascenseur muco-ciliaire et la toux, sélection d’origine immunitaire ou du fait des facteurs abiotiques locaux, compétition entre micro-organismes).
Tous ces facteurs sont autant d’éléments qui expliquent les caractéristiques bien particulières du microbiote pulmonaire bactérien que l’on peut résumer en 3 points :
(1) Une faible densité bactérienne estimée de l’ordre de 5 log par mL dans les LBA chez l’adulte et de l’ordre de 20 à 1200 bactéries pour 1000 cellules humaines dans le parenchyme pulmonaire (Hilty 2010, Sze 2012); de ce fait, l’analyse du microbiote pulmonaire par les techniques moléculaires hautement sensibles nécessite une vigilance particulière vis-à-vis des risques de contamination qui sont nombreux et justifient l’ajout systématique de témoins négatifs afin de traquer les séquences du bruit de fond (« contaminome ») (Marsh 2018).
(2) Une forte biodiversité. Si la richesse en taxons bactériens du microbiote pulmonaire est bien moindre que celle du microbiote intestinal, sa diversité est en revanche bien supérieure (alpha-diversité mesurée par l’indice de Shannon 3,3 versus 2,5 pour l’intestin) (Kim 2017) ; ceci pourrait expliquer sa capacité de résilience importante.
(3) Une grande abondance en bactéries anaérobies strictes. Les Bacteroidetes constituent ainsi près de 50% de la diversité bactérienne totale avec notamment les genres Prevotella, Veillonella, Fusobacterium ou Porphyromonas qui constituent véritablement un cœur anaérobie, pour lequel le terme « anaérobiome » a été proposé (Guilloux 2018, Lamoureux 2019). L’autre phylum principal est celui des Firmicutes, suivi dans une moindre proportion, des Proteobacteria et Actinobacteria (Charlson 2011, Dickson 2017).
Le microbiote pulmonaire non bactérien
En plus des bactéries, tous les autres domaines du vivant ont été décrits au sein de l’écosystème pulmonaire. Il a ainsi été rapporté une colonisation par des micromycètes (mycobiote) (Hunderhill 2014), des virus (virus eucaryotes et bactériophages regroupés sous le terme de virome) (Billard 2017). La présence d’Archées (archéome) est également suspectée (Koskinen 2017). Bien que ces micro-organismes prennent pleinement part au microbiote pulmonaire, peu de données sont disponibles. Avec le développement des méthodes dites de métagénomique globale par séquençage shotgun qui consistent à analyser tous les fragments ADN présents dans un échantillon et non un seul fragment comme le 16S bactérien, les connaissances sur la présence, les interactions et le rôle de ces autres micro-organismes devraient s’étendre dans les prochaines années, en particulier dans le contexte actuel de la pandémie de COVID-19 qui a permis la constitution de biocollections importantes de prélèvements respiratoires.
Chez le sujet sain, le mycobiote pulmonaire est essentiellement composé des genres Aspergillus, Cladosporium, ou Saccharomyces (Underhill 2014, Nguyen 2015). L’un des principaux réservoirs est l’air ambiant qui contient un grand nombre de spores de ces champignons ubiquistes. Certains auteurs ont montré que la composition fongique du microbiote intestinal est plus variable dans le temps que la part bactérienne de ce dernier (Dollive 2013). Il pourrait en être de même pour le microbiote pulmonaire. Le mycobiome semble être particulièrement influencé par le régime alimentaire, l’environnement et le statut immunitaire du sujet (Marsland 2014).
Le virome pulmonaire est composé majoritairement de bactériophages infectant les bactéries du microbiote pulmonaire comme Pseudomonas, Staphylococcus ou Streptococcus, ainsi que de virus infectant les cellules eucaryotes. On retrouve notamment les Picornaviridae, Paramyxoviridae, Adenoviridae, Herpesviridae, Anelloviridae, Polyomaviridae, Influenzavirus, Coronavirus, Bocavirus et Papillomavirus (Wylie 2015). La composition du virome est variable en fonction des individus et très sensible à la saisonnalité (Billard 2010).
La quantité d’Archées pulmonaires serait bien inférieure à celles de l’intestin et de la peau. Cinq phyla pulmonaires ont été identifiés : Euryarcheota, Thaumarchaeota Diapherotrites, Woesarchaeota et Aenigmarchaeota (Koskinen 2017).
Un microbiote primordial pour l’homéostasie pulmonaire
Quatre grandes catégories de fonction peuvent êtres imputées au microbiote pulmonaire.
- Rôle dans l’architecture pulmonaire. Chez les souris axéniques, des modifications anatomiques ont été notées, notamment une plus grande finesse des parois alvéolaire et capillaire. Après colonisation avec un microbiote pulmonaire, les souris initialement axéniques ont présenté une plus forte surface d’échanges alvéolaires (densité alvéolaire supérieure avec des alvéoles plus petites mais plus nombreuses) (Yun 2014).
- Rôle barrière. Ce rôle fait référence au concept d’exclusion compétitive bien décrit pour le microbiote intestinal. Il a ainsi été montré que des souris axéniques, plus sensibles aux infections pulmonaires à Streptococcus pneumoniae et Klebsiella pneumoniae, retrouvaient une résistance aux infections après transfert de microbiote par instillation nasale 72 heures avant l’infection (Brown 2017).
- Rôle dans l’immunité innée et adaptative. Ce rôle est complémentaire du précédent dans l’infection pulmonaire. Le microbiote pulmonaire a en effet également un rôle prépondérant dans l’afflux local et le pouvoir bactéricide des polynucléaires neutrophiles (Karmarkar 2013). Il est par ailleurs possible d’obtenir chez une souris saine un phénotype hyper-inflammatoire pulmonaire par simple instillation nasale du microbiote pulmonaire d’un modèle murin de BPCO (Yadava 2016). Par ailleurs, le rôle du microbiote respiratoire (en particulier du microbiote nasopharyngé) est également évoqué dans les infections virales (Pichon 2017). Ces données prennent une résonance particulière lorsque l’on évoque les pneumopathies à SARS-CoV-2.
- Rôle dans l’éducation du système immunitaire. Les études épidémiologiques suggèrent le rôle du microbiote pulmonaire dans la maturation du système immunitaire, montrant par exemple que les enfants grandissant dans un milieu urbain et exposés à un environnement très aseptisé la première année de vie sont plus à risque de développer un asthme allergique (Lynch 2014). Les bactéries anaérobies strictes ont un rôle crucial dans l’éducation du système immunitaire du poumon. Le rôle de Bacteroidetes dans la tolérance immunitaire pulmonaire a été décrit dans un modèle murin d’asthme allergique (Gollwitzer 2014). Lors des premières semaines de vie, le microbiote pulmonaire des souriceaux s’enrichit en Bacteroidetes. L’apparition de ces bactéries anaérobies strictes est corrélée à l’émergence de lymphocytes T régulateurs ainsi que de son ligand PD-L1 sur CD11b+ DC PD-L1. Ce phénomène permettrait d’induire une tolérance aux allergènes. A l’inverse, des perturbations précoces lors de la maturation du microbiote pulmonaire semblent retentir sur l’éducation du système immunitaire local et accroître le risque ultérieur d’allergie.
Un microbiote d’intérêt en médecine
Chez l’Homme, les études fonctionnelles de l’impact du microbiote pulmonaire du sujet sain sont limitées. Le rôle du microbiote respiratoire dans l’homéostasie pulmonaire a surtout été mis en évidence à travers certaines maladies chroniques à composante inflammatoire et infectieuse comme la mucoviscidose, la BPCO, l’asthme, mais également dans certaines maladies auto-immunes présentant une atteinte pulmonaire (ex. polyarthrite rhumatoïde). Toutes ces pathologies, bien que différentes dans leur symptomatologie et leur étiologie, ont en commun d’avoir l’interaction hôte – microbiote – environnement au cœur de leur physiopathologie (von Mutius 2018). De manière remarquable, ces pathologies, ont un dénominateur commun : la dysbiose pulmonaire. Ce déséquilibre se traduit le plus souvent par une augmentation de l’abondance des Proteobacteria au détriment des Bacteroidetes dont l’importance dans la fonction respiratoire est de nouveau soulignée par ces données (Marsland 2014, Caverly 2019).
Le microbiote intestinal, un partenaire clé
Les manifestations respiratoires sont fréquentes dans les maladies inflammatoires chroniques de l’intestin. A l’inverse, l’effet protecteur du microbiote digestif est reconnu dans les infections respiratoires (Samuelson 2015). Ces échanges entre les deux compartiments microbiens se feraient par voie directe ou indirecte (Marsland 2015). Dans le premier scénario, le microbiote intestinal influencerait l’écosystème pulmonaire par ensemencement direct de bactéries d’origine intestinale au niveau pulmonaire (Hoen 2015). Dans le deuxième scénario, ce serait par la production de métabolites et/ou de molécules immunomodulatrices. Le microbiote intestinal aurait une action à distance sur l’écosystème pulmonaire potentiellement dépendante de l’hématopoièse (Trompette 2014, Marsland 2015). On comprend ainsi que toute dysbiose intestinale pourrait avoir des conséquences sur l’homéostasie pulmonaire. De ce fait, la modulation du microbiote digestif (via la nutrition et les pré- ou probiotiques) pourrait constituer un levier sur la santé respiratoire comme l’a récemment prouvé un essai randomisé contrôlé réalisé sur plus de 4000 enfants (Panigrahi 2017).
Un jeune microbiote plein d’avenir
L’étude du microbiote pulmonaire fournit des nouveaux paramètres mesurables (signatures microbiennes, diversité) qui pourraient servir de biomarqueurs dans différentes maladies broncho-pulmonaires, dans le diagnostic de maladies multifactorielles complexes, dans l’identification des patients candidats à certaines thérapies (ex. corticoïdes dans l’asthme) (Goleva 2013), dans le suivi de l’effet de nouvelles thérapies (ex. correcteur du CFTR dans la mucoviscidose) (Bernarde 2015), dans le suivi de l’évolution des maladies respiratoires chroniques (Zhao 2012), dans le pronostic du risque infectieux (Keravec 2019) ou de survenue d’exacerbations (Cuthbertson 2016). Au total, l’étude du microbiote pulmonaire offre des pistes très intéressantes pour la médecine des 4P[5] (Figure 2).
Les connaissances acquises sur le microbiote pulmonaire et ses liens avec le microbiote intestinal permettent d’envisager des potentialités thérapeutiques à travers l’utilisation des probiotiques (Holt 2015). Le décryptage du microbiote pulmonaire permet également d’envisager la découverte de probiotiques de nouvelle génération. Enfin, les connaissances acquises sur l’axe intestin-poumon posent également la question d’ouvrir les indications de la transplantation de microbiote fécal aux pathologies respiratoires. Seuls des essais randomisés contrôlés permettront d’avancer sérieusement sur ces nouvelles pistes thérapeutiques.
En conclusion, l’étude du microbiote pulmonaire offre des perspectives prometteuses en termes de clés physiopathologiques des maladies respiratoires multifactorielles et de nouvelles potentialités (diagnostiques, pronostiques, thérapeutiques) parfaitement en ligne avec le développement de la médecine de précision.
[1] Pneumonie franche lobaire aiguë
[2] Bronchopneumopathie chronique obstructive
[3] National Institutes of Health
[4] Lavage broncho-alvéolaire
[5] Prédictive, préventive, personnalisée et participative

Figure 1. Première de couverture des Archives de Médecine Expérimentale et d’Anatomie Pathologique, revue dans laquelle l’article décrivant la présence de bactéries dans les poumons de cadavres exempts de toute affection pulmonaire fut publié en 1899 (source : gallica.bnf.fr).
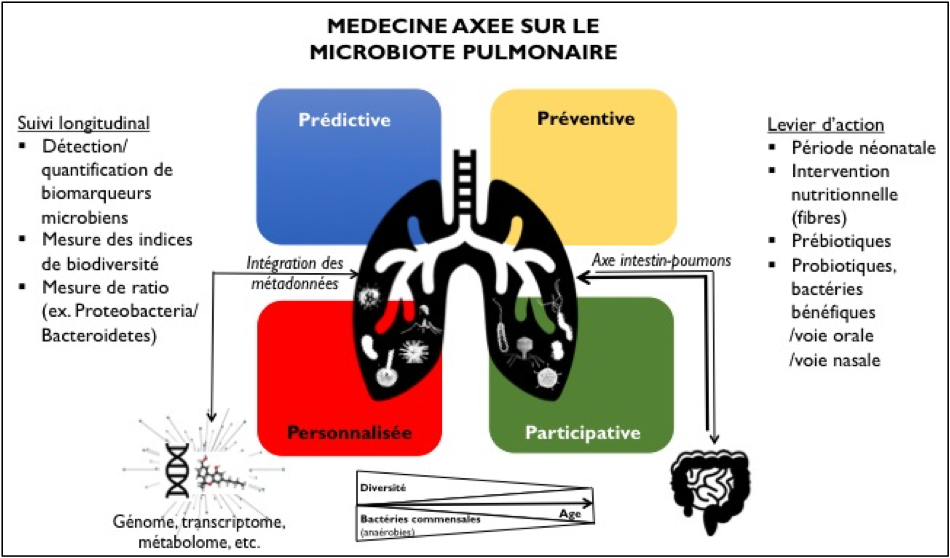
Figure 2. Le microbiote pulmonaire comme outil de la médecine des 4P pour l’amélioration de la santé respiratoire. D’après l’OMS, plus de 300 millions de personnes souffriraient de pathologies respiratoires chroniques dans le monde.
Pr Geneviève Héry-Arnaud
UFR Médecine / INSERM/ CHRU Brest
Service de Bactériologie-Virologie / UMR1078 « Axe Microbiota »
Genevieve.Hery-Arnaud@univ-brest.fr
Bibliographie