Objectif
Etudier la distribution du signal RT-PCR (Ct) sur les échantillons détectés positifs pour le SARS-CoV-2. Analyser dans quelles proportions un test antigénique aurait rendu un résultat faussement négatif du fait d’un signal faible en PCR (Ct élevé).
Technique utilisée, hétérogénéité des demandes de diagnostics et analyse
La trousse de détection par RT-PCR est celle de Thermofisher “TaqPathTM COVID-19 CE-IVD RT-PCR V2” utilisée sur un QuantStudioTM 5 System.
En se référant au rapport de la SFM sur la comparaison des différentes trousses [Avis du 25 septembre 2020 de la Société Française de Microbiologie (SFM) relatif à l’interprétation de la valeur de Ct], on note que “l’excrétion virale modérée voire très faible” est définie par des Ct supérieurs à 28 ou 32, respectivement. Ces valeurs sont sensiblement plus faibles avec cette technique que celles obtenues avec la technique de référence de l’institut Pasteur, 33 et 37, respectivement.
L’analyse a été effectuée en utilisant les valeurs de Ct du gène N uniquement; cible la plus fréquemment détectée parmi les 3 gènes ciblés par le test (ORF1ab, N et S).
L’analyse globale prend en compte tous les échantillons détectés positifs. Le recrutement au CHU est très hétérogène : “Drive” divers, CH de la région Bretagne, dépistage du personnel et patients hospitalisés au CHU. Quelques analyses ciblées sont proposées pour des populations identifiées.
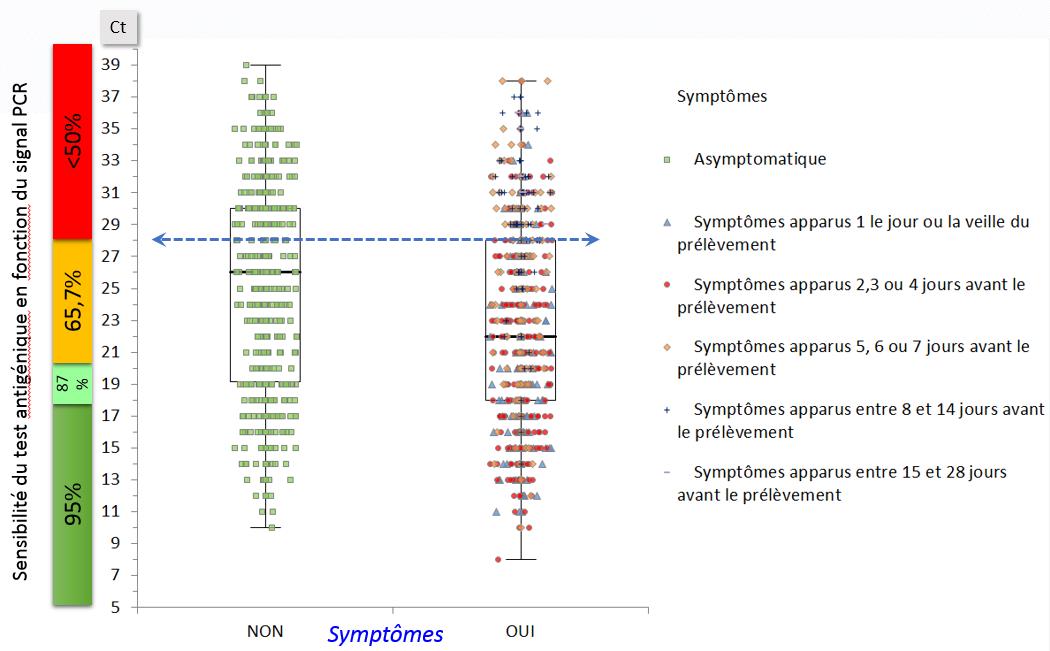
L’analyse en fonction des symptômes ne prend en compte que le Drive sur site et la notion de délai des symptômes est issu d’un questionnaire rempli par le patient. La validité de la déclaration doit donc être considérée avec recul.
L’analyse “virtuelle” par rapport au test antigénique est proposée par rapport aux données du rapport d’Henri Mondor sur les tests antigéniques avec un seuil à 28 et 32 pour notre technique PCR et en prenant comme modèle le test Abbott.
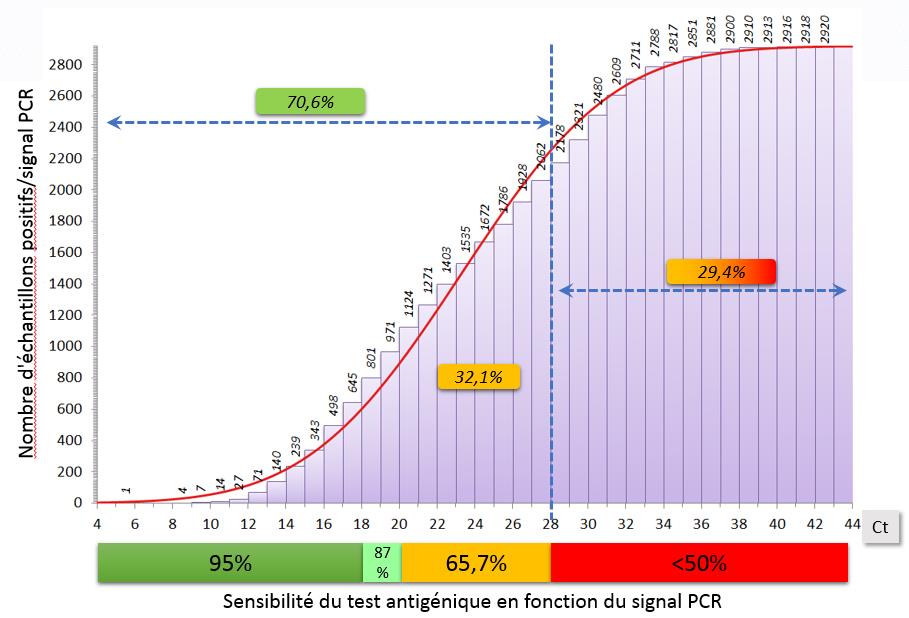
Distribution du signal PCR (Ct) sur 2920 résultats positifs
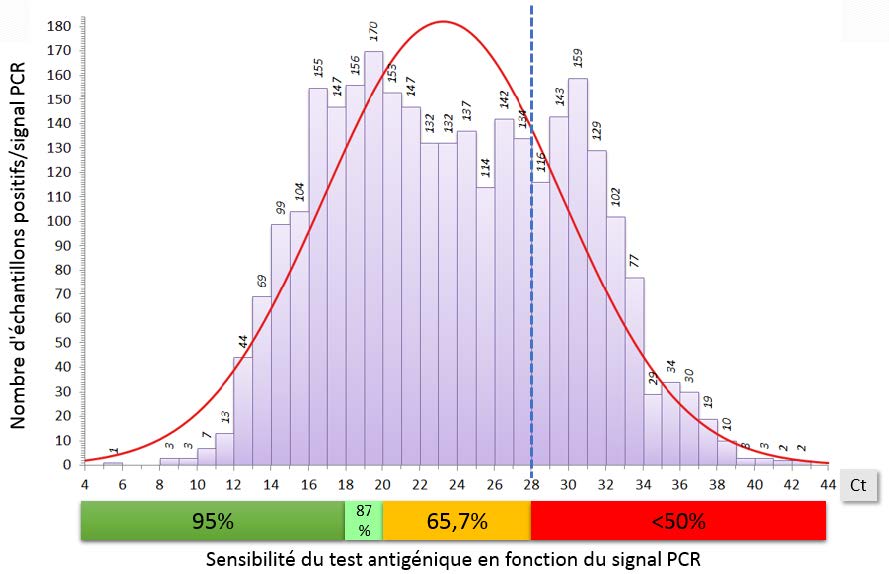
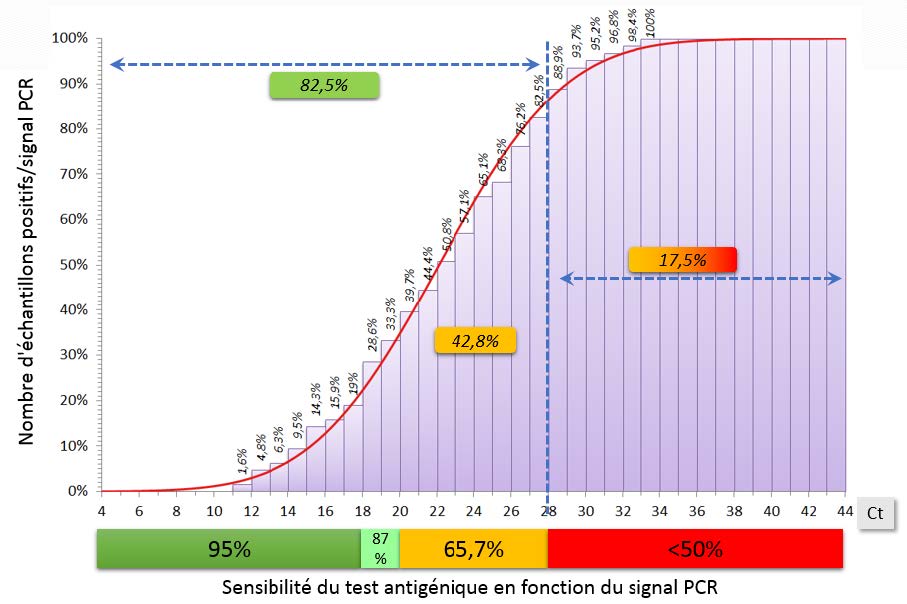
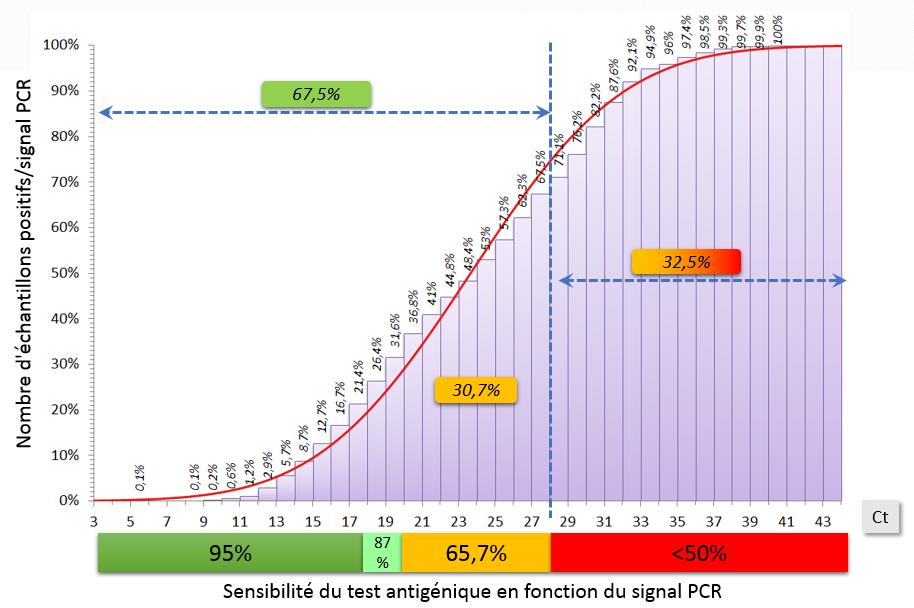
Pourcentage d’échantillons distribués selon la sensibilité du test antigénique
Analyse sur 66 prélèvements prélevés au service des urgences
Analyse sur 1362 prélèvements prélevés dans un contexte de drive
Analyse sur 1303 prélèvements avec une information concernant les symptômes
Conclusion
Cette analyse et ces représentations permettent d’objectiver le risque de rater un diagnostic en utilisant un test antigénique par rapport à l’utilisation d’un test moléculaire dans des conditions réelles de diagnostic.
La simulation a été effectuée en prenant comme exemple les sensibilités décrites pour le test Abbott et en se plaçant dans des situations comme un dépistage “tout-venant” dans un Drive ou pour un diagnostic aux urgences.
Informations partagées par l’équipe de Service de Virologie du CHU de Rennes
Contact :
Pr Vincent THIBAULT
Vincent.THIBAULT@chu-rennes.fr
Service de Virologie – CHU Rennes
Université Rennes / INSERM / EHESP / IRSET – UMR_S 1085






