Staphylococcus xylosus une bactérie bien adaptée à la croûte de fromage
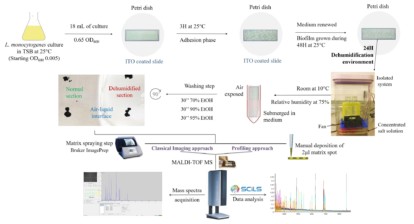
Staphylococcus xylosus fait partie du microbiote des fromages traditionnels. Il est particulièrement présent dans le microbiote de surface qui contribue aux propriétés sensorielles des fromages. Malgré sa fréquence, les mécanismes moléculaires permettant sa croissance et son adaptation dans les produits laitiers sont encore mal connus. Une approche transcriptomique a été utilisée pour déterminer comment le profil d’expression génique est modifié pendant l’étape de fermentation dans une matrice laitière solide. S. xylosus développe un métabolisme aérobie adapté à la surface du fromage. Il surexprime des gènes impliqués dans le catabolisme aérobie de deux sources de carbone de la matrice laitière, le lactose et le citrate. Fait intéressant, S. xylosus doit faire face à une pénurie nutritionnelle en acides aminés et nucléotides, et par conséquent, une surexpression des gènes impliqués dans leur biosynthèse est observée. Comme attendu, le gène sigB est surexprimé en rapport au stress général lié à l’entrée en phase stationnaire de la bactérie et plusieurs gènes sous sa régulation, tels que ceux impliqués dans le transport des anions, des cations et dans la pigmentation sont surexprimés. La surexpression des gènes codant pour les enzymes antioxydantes et les systèmes de transport et de synthèse de la glycine-bétaïne révèle que S. xylosus doit faire face à un stress oxydatif et osmotique. De plus, S. xylosus exprime un système original potentiellement impliqué dans l’acquisition du fer à partir de la lactoferrine.
Cette étude apporte des connaissances qui pourront être utilisées pour analyser les données métatranscriptomiques de l’ensemble de la communauté microbienne d’affinage des fromages.
Contact : Sabine Leroy, sabine.leroy@inrae.fr
Pour en savoir plus :
Sabine Leroy, Sergine Even, Pierre Micheau, Anne de La Foye, Valérie Laroute, Yves Le Loir, Régine Talon. Transcriptomic Analysis of Staphylococcus xylosus in Solid Dairy Matrix Reveals an Aerobic Lifestyle Adapted to Rind. Microorganisms 2020, 8, 1807; doi:10.3390/microorganisms8111807