Depuis 1976, plus d’une trentaine d’épidémies de maladie à virus Ebola (MVE) ont été détectées en Afrique sub-Saharienne, dont 8 depuis 2013, dernière année sans épidémie de MVE. Les deux plus grandes épidémies ont frappé l’Afrique de l’Ouest, et en particulier la Guinée, le Liberia et la Sierra Leone, de 2014 à 2016, et l’Est de la République Démocratique du Congo de 2018 à 2020, faisant respectivement 11343 et 2299 victimes.
Etant donné l’issue très souvent fatale de l’infection par le virus Ebola, il est généralement admis que celui-ci n’infecte pas l’être humain de manière endémique. Les flambées épidémiques correspondraient ainsi plutôt à des évènements d’émergence zoonotique. Les chauves-souris sont les candidates les plus sérieuses au statut de réservoir mais la question est encore loin d’être définitivement tranchée. Chaque nouvelle épidémie constitue une occasion importante de mieux comprendre l’écologie et le processus d’émergence du virus Ebola.
En mars 2021, une deuxième épidémie d’Ebola était déclarée en Afrique de l’Ouest, en Guinée. Cette épidémie allait affecter au moins 16 personnes (cas confirmés), et en tuer 12. Les autorités sanitaires locales, épaulées par plusieurs équipes internationales, ont pu reconstruire la chaîne des infections jusqu’à un cas index plausible, une infirmière probablement décédée de la maladie à la fin du mois de janvier.
Comme c’est désormais devenu la norme, le séquençage sur site, et en quasi temps réel, de génomes viraux fut réalisé. Les premières séquences partielles confirmèrent que des 4 espèces de virus pouvant causer la MVE, celle impliquée était aussi celle contre laquelle des vaccins et traitements efficaces étaient disponibles, permettant leur mise en œuvre rapide.
L’analyse des génomes complets allait révéler une information bien plus surprenante. Ceux-ci semblaient en effet dériver directement de la précédente épidémie Ouest-africaine, pourtant terminée en 2016. On le voit clairement dans un arbre phylogénétique représentant les relations de parenté des virus Ebola de cette espèce: les séquences de l’épidémie de 2021 sont toutes nichées au sein de la diversité virale générée entre 2014 et 2016 (Figure 1).
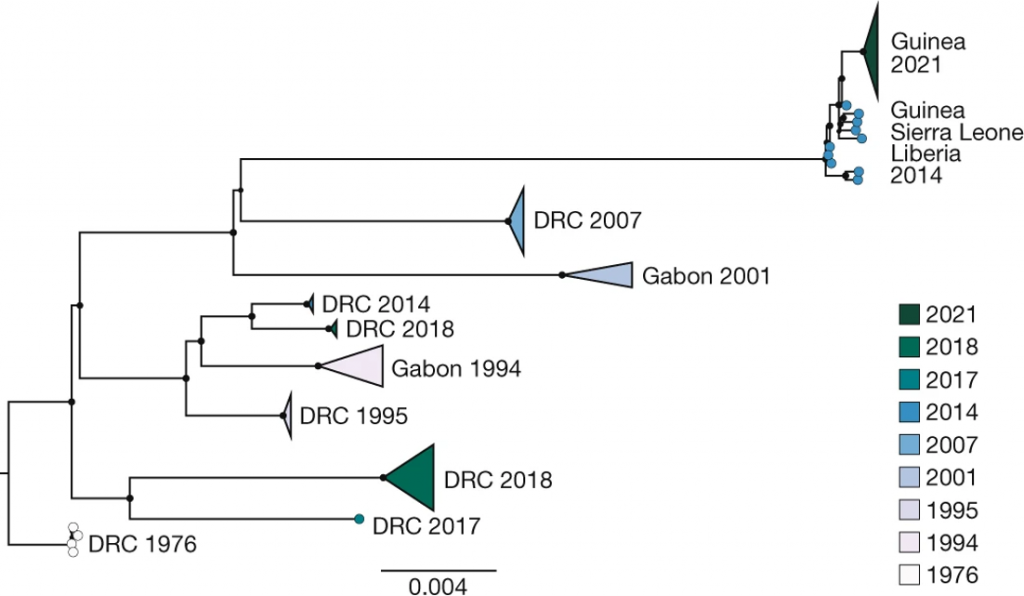
Autre conclusion étonnante: la branche de l’arbre menant aux virus de 2021 est bien plus courte qu’attendue. En appliquant des modèles d’horloge moléculaire (dont le ‘tic-tac’ représente l’accumulation plus ou moins régulière des mutations au cours du temps), un ralentissement du taux d’évolution d’un facteur 6 peut être détecté. À vrai dire, lorsqu’on demande à ces mêmes modèles d’estimer l’âge des séquences de 2021, ils suggèrent la date de 2015, les replaçant au cœur de l’épidémie précédente.
Ces résultats indiquent clairement que l’épidémie de 2021 ne fait pas suite à une nouvelle émergence zoonotique mais représente plutôt une résurgence de celle de 2014-6. Un des mécanismes envisageables est une latence ou dormance du virus dans des sites anatomiques immunoprivilégiés; on sait par exemple que le virus peut persister des mois dans le sperme de patients guéris. On sait désormais également que des résurgences similaires ont eu lieu lors d’autres épidémies, par exemple lors de celle du Nord-Kivu en 2018-20. L’épidémie de 2021 en Guinée démontre cependant que de tels évènements peuvent se produire bien plus longtemps après la fin d’une précédente épidémie que ce que nous pensions jusqu’alors.
Environ 20000 personnes ont survécu aux épidémies de MVE au cours des 10 dernières années. La découverte des résurgences de MVE montre que le suivi de ces patients est absolument crucial pour prévenir de futures épidémies. Ce suivi devra comprendre des mesures d’accompagnement adaptées, pour éviter que ces populations déjà très fragilisées ne soient encore davantage stigmatisées.
Sebastien Calvignac-Spencer, Ph.D.
Epidemiology of Highly Pathogenic Microorganisms Viral Evolution Robert Koch Institut
CalvignacS@rki.de
www.leendertz-lab.org www.pathogen-museomics.org
Lire l‘article source :
Keita AK, Koundouno FR, Faye M, Düx A, Hinzmann J, Diallo H, Ayouba A, Le Marcis F, Soropogui B, Ifono K, Diagne MM, Sow MS, Bore JA, Calvignac-Spencer S, Vidal N, Camara J, Keita MB, Renevey A, Diallo A, Soumah AK, Millimono SL, Mari-Saez A, Diop M, Doré A, Soumah FY, Kourouma K, Vielle NJ, Loucoubar C, Camara I, Kourouma K, Annibaldis G, Bah A, Thielebein A, Pahlmann M, Pullan ST, Carroll MW, Quick J, Formenty P, Legand A, Pietro K, Wiley MR, Tordo N, Peyrefitte C, McCrone JT, Rambaut A, Sidibé Y, Barry MD, Kourouma M, Saouromou CD, Condé M, Baldé M, Povogui M, Keita S, Diakite M, Bah MS, Sidibe A, Diakite D, Sako FB, Traore FA, Ki-Zerbo GA, Lemey P, Günther S, Kafetzopoulou LE, Sall AA, Delaporte E, Duraffour S, Faye O, Leendertz FH, Peeters M, Toure A, Magassouba NF. Resurgence of Ebola virus in 2021 in Guinea suggests a new paradigm for outbreaks. Nature. 2021 Sep;597(7877):539-543. doi: 10.1038/s41586-021-03901-9. Epub 2021 Sep 15. PMID: 34526718.
https://pubmed.ncbi.nlm.nih.gov/34526718/