Comparaison des séquences génomiques pour la surveillance épidémiologique et l’investigation d’épidémies : 3 approches complémentaires
François Gravey1, 2 et Sylvain Brisse3
- CHU de Caen, Service de bactériologie, Caen, France
- DynaMicURe – Dynamique Microbienne associée aux Infections Urinaires et Respiratoires – UMR 1311 –Université de Caen, Université de Rouen, Inserm
- Institut Pasteur, Université Paris Cité, Unité Biodiversité et Epidémiologie des Bactéries Pathogènes
Depuis les années 2010, la capacité des nouveaux séquenceurs dits de ‘nouvelle génération’ a considérablement réduit la durée et les coûts de production des données génomiques 1. Le séquençage du génome complet des micro-organismes est ainsi devenu largement accessible aux laboratoires de microbiologie clinique et d’hygiène hospitalière. En janvier 2023, la base de données NCBI comptait 507 920 génomes d’organismes procaryotes, 56625 génomes de virus et 27755 génomes de champignons (https://www.ncbi.nlm.nih.gov/genome/microbes/).
Avec cette possibilité nouvelle viennent des défis nouveaux : analyser les données pour répondre à des questions de transmission des pathogènes, de source des infections, et de leurs caractéristiques médicalement pertinentes, comme la résistance aux antibiotiques 2–4. Face à une telle masse d’informations, nombreux sont également les projets de recherche ayant pour objectif d’analyser les différentes populations de micro-organismes. Afin de rendre cela possible, plusieurs approches bio-informatiques ont été développées ; l’objet de cet article est de présenter les différentes méthodes actuellement disponibles pour comparer les souches entre elles et étudier les populations microbiennes.
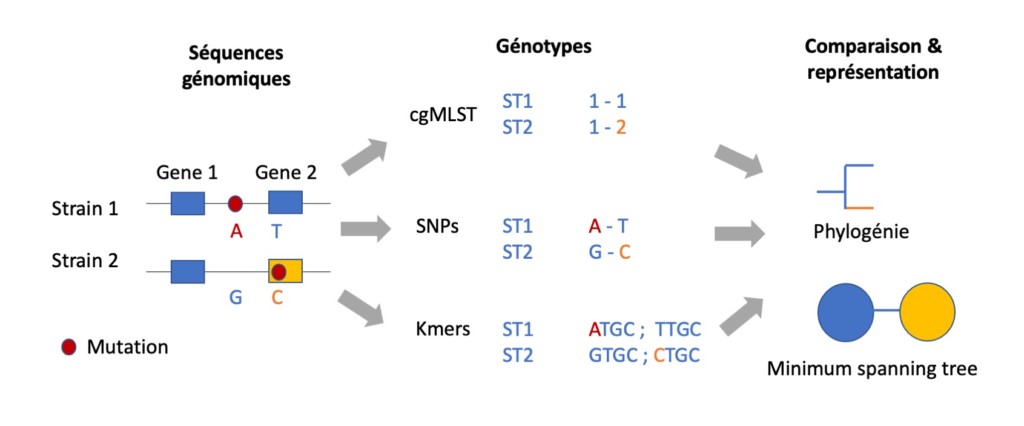
Il existe trois grands types d’approches. La première s’appuie sur des séquences de référence contre lesquelles les génomes étudiés seront alignés et comparés. La seconde est l’approche gène-par-gène, dite Multi-Locus Sequence Typing (MLST) ou son extension, le core genome Multi-Locus Sequence Typing (MLST) ; elle repose sur un sous-ensemble de marqueurs génétiques prédéfinis, typiquement les gènes en copie unique dans les génomes. Enfin, la troisième approche ne nécessite ni référence ni marqueurs génétiques prédéfinis : elle étudie les génomes deux à deux en comparant leur contenu en ‘mots’ c’est-à-dire en séquences nucléotidiques d’une longueur ‘k’ choisie, appelés k-mers.
Cet article présentera les principes fondamentaux de ces trois approches, qui sont schématisés sur la Figure. Il sera suivi de deux autres articles qui viendront illustrer comment ces méthodes sont utilisées en surveillance et épidémiologie génomique et en recherche en biologie des populations et taxonomie des souches.
Approches MLST et dérivées : core-genome MLST
L’approche MLST dite classique repose sur l’analyse d’un petit nombre de gènes de ménage, typiquement 7 gènes. Chaque variant de séquence d’un gène est codée en un numéro d’allèle. La combinaison des allèles identifiés au sein d’une souche est résumée par un identifiant unique, le « séquençotype » (ST, sequence type en anglais). Le premier schéma MLST a été créé en 1998 dans le but de caractériser des souches de Neisseria meningitidis pour lesquelles les méthodes de typage phénotypiques se révélaient décevantes 5. Un schéma MLST a maintenant été défini pour chaque espèce pathogène (ou même d’autres espèces, environnementales ou probiotiques) d’importance. Ces schémas MLST et les variants des séquences répertoriées sont disponibles au sein de différentes bases de données accessibles publiquement dont les principales sont : PubMLST (https://pubmlst.org/), BIGSdb-Pasteur (https://bigsdb.pasteur.fr/), EnteroBase (https://enterobase.warwick.ac.uk/), Chewie-NS (https://chewie-ns.readthedocs.io/en/latest/) ou le site cgmlst.org (https://cgmlst.org/ncs) maintenu par la compagnie SeqSphere.
L’avantage du MLST est de fournir des génotypes standardisés, portables, compréhensibles facilement. L’inconvénient des systèmes MLST classiques, créés pour le séquençage Sanger, est de reposer sur un petit nombre de gènes qui représentent typiquement à peine 1/1000e de la taille du génome. Ces approches ont toutefois rendu un énorme service en fournissant une nomenclature des principales lignées phylogénétiques (ou groupes clonaux) au sein des espèces bactériennes ; ces taxonomies de souches se sont imposées quasi-universellement, sauf chez les pathogènes monomorphes chez lesquels la diversité génétique est insuffisante, comme Mycobacterium tuberculosis ou le sérotype Typhi de Salmonella par exemple.
Les développements technologiques de séquençage haut débit associés aux progrès des outils bio-informatiques ont permis d’obtenir avec plus de facilité des génomes quasi-complets. Le contenu en gènes s’est révélé très variable entre souches d’une même espèce. Ainsi, par convention, le « core genome » est défini comme la liste des gènes conservés (définis par exemple comme étant présents au sein de 95% des génomes étudiés) tandis que le « pangenome » est l’ensemble des gènes retrouvés au moins une fois. Le core genome et le pangénome sont typiquement définis pour une espèce, ou plus généralement pour tout ensemble de génomes étudiés.
La définition de « core genomes » a permis d’appliquer l’approche MLST classique à plus grande échelle. L’inclusion de core genes dans les schémas cgMLST dépend des propriétés de discrimination ou d’éventail d’applicabilité recherchés. A titre d’illustration, le schéma « core genome MLST » de Escherichia coli contient 2513 gènes pour maximiser la discrimination entre souches de cette espèce, tandis que celui de Klebsiella pneumoniae contient 629 gènes conservés non seulement dans K. pneumoniae sensu stricto mais dans les espèces proches (K. pneumoniae species complex, KpSC) 6,7.
Dans cette approche dite gène par gène, chaque souche étudiée se voit attribuer une suite de numéros correspondant aux allèles retrouvés dans chaque gène (locus) du schéma utilisé (Figure). Une estimation de la distance génétique entre deux souches est fournie par le nombre d’allèles différents. A noter que cette distance allélique est une approximation qui ne laisse pas présager du nombre exact de modifications nucléotidiques observées entre les génomes. L’ensemble des distances deux-à-deux est résumé dans une matrice de distance, qui sert à visualiser les relations de proximité génétique sous la forme d’un regroupement hiérarchique ou d’un « minimum-spanning tree », approche rendue très populaire par des outils comme GrapeTree 8–10.
En résumé, les méthodes MLST ne dépendent pas d’un génome de référence et s’appuient sur un schéma (ensemble de marqueurs génétiques) standard lié à une base de données publique qui permet un inventaire de la diversité et la comparaison inter-laboratoires des génomes 6,8,11. Une différence notable entre MLST classique et cgMLST est, pour cette seconde approche, la tolérance de données manquantes. En effet, les assemblages incomplets des séquences génomiques, ou des évènements évolutifs de perte de gènes, peuvent résulter en l’absence de certains gènes (loci) du schéma cgMLST. Il est donc important de pouvoir accepter des données manquantes dans l’approche cgMLST, contrairement au MLST qui repose typiquement sur des gènes indispensables au fonctionnement cellulaire. La proportion de données manquantes, que l’on cherche toutefois à minimiser, est un critère important lors de la création de schémas cgMLST. L’existence de quelques données manquantes apporte une certaine imprécision à la comparaison des génomes 7 mais apporte la souplesse indispensable à la définition de génotypes (cgST, pour core genome Sequence Type) selon cette approche. L’approche cgMLST est encore largement en développement mais des schémas cgMLST largement utilisés sont déjà disponibles publiquement pour les principaux pathogènes, par exemple Listeria monocytogenes, K. pneumoniae, Salmonella enterica, E. coli, Campylobacter ou Neisseria 6,7,12–14.
Une propriété très importante qui a fait le succès de l’approche MLST est d’être le socle de nomenclatures des variants bactériens. L’approche MLST classique a défini des sequençotypes (ST) qui sont largement utilisés comme taxonomie internationale des souches. L’approche cgMLST peut également être utilisée pour définir des génotypes, lignées ou groupes clonaux. Ces aspects de classification et nommage des variants feront l’objet d’un article ultérieur.
Étude des variations nucléotidiques sur l’ensemble du génome : Insertion, Délétion et Single Nucleotide Polymorphisms
Une autre approche consiste à détecter au sein des génomes les variations de séquence nucléotidique par rapport à une séquence de référence. Différentes modifications peuvent être détectées : les insertions qui correspondent à quelques nucléotides surnuméraires par rapport au génome de référence, les délétions qui sont des pertes de nucléotides et enfin des mutations ponctuelles résultant en des polymorphismes nucléotidiques simples appelés par leur dénomination en anglais : Single Nucleotide Polymorphism (SNP) 15.
Ces analyses sont réalisées via alignement (mapping, terme anglais plus utilisé) des reads (lectures individuelles des séquences d’ADN issues du séquenceur). Ces analyses nécessitent l’utilisation des fichiers au format « fastq », qui contiennent toutes les séquences issues du séquenceur, et des informations sur leur qualité. Ces fichiers peuvent contenir des millions de séquences individuelles et sont typiquement très volumineux ; leur manipulation nécessite donc des moyens informatiques dédiés.
La première étape de la recherche de variations consiste, après une étape de contrôle qualité des séquences, à aligner une par une les séquences individuelles issues du séquenceur, contre le génome de référence. Plusieurs outils sont disponibles pour réaliser cette étape de mapping, dont BWA et bowtie2 16–18. Une fois cette étape terminée, chaque nucléotide de la séquence de référence est couvert par différents reads qui y ont été alignés, définissant la profondeur du mapping. Cette notion de profondeur est très importante car elle permet de discriminer les erreurs de séquençage aléatoires des « vrais variants » présents dans la souche étudiée. En effet, si une observation est issue d’une erreur aléatoire, peu de reads porteront cette information (la profondeur du variant sera faible) tandis que pour un « authentique SNP », quasiment tous les reads porteront la mutation (la profondeur du variant sera importante)
Une seconde étape dite de variant calling va alors dresser la liste de toutes les modifications génomiques retrouvées entre le génome de référence et la souche étudiée. Ce processus utilise différentes métriques dont : la qualité du séquençage de la base (le score Phred), la qualité de l’alignement des reads et leur concordance, qui tient compte de la profondeur du mapping. Il existe différents algorithmes qui permettent le variant calling dont ceux des outils FreeBayes, GATK ou SAMtools 19–21. L’ensemble des variations génomiques retrouvées est enregistré dans un fichier appelé Variant Call Format (VCF). De façon très intéressante, si la séquence de référence est annotée, il est possible de connaitre quelles régions intergéniques ou quels gènes sont modifiés dans la souche étudiée, et de s’intéresser aux conséquences vis-à-vis de leur expression et fonctionnalité.
Dans une troisième étape, la comparaison des variations trouvées permet d’estimer la ressemblance entre génomes. Dans certaines approches, seules les bases du génome de référence couvertes par toutes les souches étudiées sont utilisées pour calculer les distances génomiques ; ceci constitue donc une approche « core-SNP ». Aussi, pour que ces analyses soient les plus puissantes et précises possibles, le génome de référence utilisé doit être de bonne qualité et phylogénétiquement proche des souches étudiées, c’est-à-dire, à moins de 1% de distance nucléotidique, donc typiquement du même ST 9. Parallèlement, la qualité des génomes comparés compte aussi pour beaucoup, car si l’une des souches présente un alignement de mauvaise qualité, c’est la comparaison de l’ensemble des génomes qui risque d’être altérée 9.
A la fin du processus, pour chaque souche étudiée, la position et le nombre des SNP par rapport à la référence sont connus. Il est alors possible de déduire la distance génomique entre les souches en étudiant position par position la présence et la nature des variants retrouvés par rapport à la référence.
Un grand avantage de cette approche est d’analyser sans a priori toutes les variations entre génomes. Cette approche maximise donc la discrimination entre souches, et permet de découvrir des variations inattendues, contrairement à l’approche MLST qui définit a priori des régions à étudier. Toutefois, la dépendance de l’approche SNP vis-à-vis d’une référence n’est pas sans inconvénients. En effet, il n’est pas possible de comparer les souches directement entre études différentes, surtout si ces dernières ont utilisé des références différentes. De plus, les régions génomiques répétées (par exemple des séquences d’insertion ou des gènes répétés, comme les opérons ribosomiques) posent problème aux outils de mapping et génèrent des variations artéfactuelles. Il convient donc de repérer ces zones répétées, ce qu’il faut faire pour chaque nouvelle référence utilisée, et de filtrer les variants issus de ces régions. Enfin, il faut tenir compte de la présence d’artéfacts de mapping éventuels non définissables a priori en cas de nouveaux clones pour lesquels il n’y a peu ou pas d’expérience avec les souches de référence.
Comparaison des mots (k-mers) contenus dans les génomes
Contrairement aux deux approches ci-dessus, l’approche « k-mers » ne dépend ni d’un génome de référence ni d’un ensemble de marqueurs génétiques préexistant. Le grand avantage des approches k-mers est donc d’être applicables universellement. Elles reposent sur l’étude du contenu en séquences nucléotidiques d’une longueur définie « k » de tous les génomes étudiés. Ainsi, pour chaque génome, un catalogue de tous les k-mers retrouvés est réalisé. Par la suite, ce sont ces contenus en k-mers qui sont comparés deux à deux et qui définissent la distance entre deux génomes d’intérêt 9.
Des approches très novatrices basées sur l’approche k-mers utilisent des techniques avancées pour comparer un grand nombre de génomes, initialement développées par des moteurs de recherche 22. Elles reposent sur l’approche MinHash qui permet de réduire une très grande quantité de données en un plus petit volume dont la gestion demande moins de ressources computationnelles 22.
L’algorithme le plus utilisé reposant sur cette approche est l’outil Mash dont le fonctionnement peut être résumé en trois étapes 23 : (i) pour chaque génome, les k-mers présents sont recensés ; (ii) chaque séquence de k-mer est réduite (« hashée ») en un identifiant unique de plus petite taille. Cette étape a pour but de diminuer la quantité d’informations qu’il faudra comparer et donc placer dans la mémoire des ordinateurs ; (iii) enfin, seul un échantillon représentatif de ces hashs sera conservé dans un contenant appelé sketch : c’est l’approche MinHash.
La comparaison de génomes via ces outils correspond donc à la comparaison du contenu en hashs de leurs sketches. Les performances de ces approches sont bonnes et permettent de différencier les isolats dans une gamme étendue de distances génétiques 22,23. Il a été démontré que les regroupements réalisés par ces outils étaient comparables à des résultats d’analyses cgMLST 9. Différents outils sont disponibles notamment PopPUNK qui a pour originalité de comparer séparément le core genome et le pangénome des souches étudiées 24.
En plus de s’affranchir de toutes les problématiques de référence, les approches k-mers utilisent l’ensemble des données nucléotidiques présentes dans les génomes ; les comparaisons ne sont donc pas réduites aux seules séquences communes des génomes étudiés.
Conclusions
Ainsi, en pratique : le cgMLST est une première approche standardisée qui permet de se repérer dans la classification des souches d’une espèce, et apporte le plus souvent une discrimination suffisante pour exclure des relations clonales. S’il existe une identité au regard du cgMLST, il convient alors d’utiliser l’approche SNP qui maximise la discrimination, ce qui est indispensable au sein d’épidémies clonales. L’approche SNP permet également de découvrir toutes les variations génétiques, sans se limiter à des marqueurs prédéfinis. Pour des jeux de données d’espèces moins connues ou très divers (plusieurs espèces) ou pour lesquelles il n’existe pas de schéma MLST ou cgMLST, ou si l’on souhaite utiliser une seule méthode pour de nombreuses espèces, les approches k-mers apportent une classification rapide des génomes qui tient compte à la fois de leur proximité phylogénétique et de leur contenu en gènes.
Références bibliographiques
-
Mardis, E. R. DNA sequencing technologies: 2006–2016. Nat. Protoc. 12, 213–218 (2017).
-
Read, T. D. et al. Comparative Genome Sequencing for Discovery of Novel Polymorphisms in Bacillus anthracis. Science 296, 7 (2002).
-
Köser, C. U. et al. Rapid Whole-Genome Sequencing for Investigation of a Neonatal MRSA Outbreak. N. Engl. J. Med. (2012).
-
Nordmann, P., Dortet, L. & Poirel, L. Carbapenem resistance in Enterobacteriaceae: here is the storm! Trends Mol. Med. 18, 263–272 (2012).
-
Maiden, M. C. J. et al. Multilocus sequence typing: A portable approach to the identification of clones within populations of pathogenic microorganisms. Proc. Natl. Acad. Sci. 95, 3140–3145 (1998).
-
Zhou, Z. et al. The EnteroBase user’s guide, with case studies on Salmonella transmissions, Yersinia pestis phylogeny, and Escherichia core genomic diversity. Genome Res. 30, 138–152 (2020).
-
Hennart, M. et al. A Dual Barcoding Approach to Bacterial Strain Nomenclature: Genomic Taxonomy of Klebsiella pneumoniae Strains. Mol. Biol. Evol. 39, msac135 (2022).
-
Schürch, A. C., Arredondo-Alonso, S., Willems, R. J. L. & Goering, R. V. Whole genome sequencing options for bacterial strain typing and epidemiologic analysis based on single nucleotide polymorphism versus gene-by-gene–based approaches. Clin. Microbiol. Infect. 24, 350–354 (2018).
-
Uelze, L. et al. Typing methods based on whole genome sequencing data. One Health Outlook 2, 3 (2020).
-
Zhou, Z. et al. GrapeTree: visualization of core genomic relationships among 100,000 bacterial pathogens. Genome Res. 28, 1395–1404 (2018).
-
Jolley, K. A., Bray, J. E. & Maiden, M. C. J. Open-access bacterial population genomics: BIGSdb software, the PubMLST.org website and their applications. Wellcome Open Res. 3, 124 (2018).
-
Moura, A. et al. Whole genome-based population biology and epidemiological surveillance of Listeria monocytogenes. Nat. Microbiol. 2, 16185 (2016).
-
Cody, A. J., Bray, J. E., Jolley, K. A., McCarthy, N. D. & Maiden, M. C. J. Core Genome Multilocus Sequence Typing Scheme for Stable, Comparative Analyses of Campylobacter jejuni and C. coli Human Disease Isolates. J. Clin. Microbiol. 55, 2086–2097 (2017).
-
Harrison, O. B. et al. Neisseria gonorrhoeae Population Genomics: Use of the Gonococcal Core Genome to Improve Surveillance of Antimicrobial Resistance. J. Infect. Dis. 222, 1816–1825 (2020).
-
den Bakker, H. C. et al. A Whole-Genome Single Nucleotide Polymorphism-Based Approach To Trace and Identify Outbreaks Linked to a Common Salmonella enterica subsp. enterica Serovar Montevideo Pulsed-Field Gel Electrophoresis Type. Appl. Environ. Microbiol. 77, 8648–8655 (2011).
-
Alser, M. et al. Technology dictates algorithms: recent developments in read alignment. Genome Biol. 22, 249 (2021).
-
Li, H. & Durbin, R. Fast and accurate short read alignment with Burrows-Wheeler transform. Bioinformatics 25, 1754–1760 (2009).
-
Langmead, B. & Salzberg, S. L. Fast gapped-read alignment with Bowtie 2. Nat. Methods 9, 357–359 (2012).
-
DePristo, M. A. et al. A framework for variation discovery and genotyping using next-generation DNA sequencing data. Nat. Genet. 43, 491–498 (2011).
-
Li, H. et al. The Sequence Alignment/Map format and SAMtools. Bioinformatics 25, 2078–2079 (2009).
-
Garrison, E. & Marth, G. Haplotype-based variant detection from short-read sequencing. Preprint at http://arxiv.org/abs/1207.3907 (2012).
-
Argimón, S. & Aanensen, D. M. Species Mash-up. Nat. Rev. Microbiol. 14, 730–730 (2016).
-
Ondov, B. D. et al. Mash: fast genome and metagenome distance estimation using MinHash. Genome Biol. 17, (2016).
-
Lees, J. A. et al. Fast and flexible bacterial genomic epidemiology with PopPUNK. Genome Res. 29, 304–316 (2019).








