La taxonomie des souches bactériennes à l’heure de la génomique
Mélanie Hennart1, Alexis Criscuolo2 et Sylvain Brisse1,2
1. Institut Pasteur, Université Paris Cité, Unité Biodiversité et Épidémiologie des Bactéries Pathogènes, F-75015 Paris, France
2. Institut Pasteur, Université Paris Cité, Centre de Ressources Biologiques de l’Institut Pasteur, F-75015 Paris, France
Introduction
Pour permettre la communication entre les différents acteurs de la surveillance épidémiologique, il est important de disposer de systèmes précis de classification et de nommage des souches bactériennes circulantes, ainsi que de leurs variants potentiellement pathogènes. La taxonomie (au sens de la systématique des organismes vivants) peut se décomposer en trois opérations complémentaires et interdépendantes : la classification, la nomenclature et l’identification. Tout comme pour les organismes eucaryotes pluricellulaires, la taxonomie procaryote est basée sur le système linnéen (i.e. phylum, classe, ordre, famille, genre, espèce, sous-espèce). Cette taxonomie ne prend donc pas du tout en compte des niveaux plus fins que la sous-espèce (phylogroupes, lignées, souches, clones …). Pourtant, des lignées phylogénétiques distinctes au sein de nombreuses (sous-)espèces microbiennes peuvent posséder des propriétés très différentes en termes d’écologie ou de pathogénicité. Un exemple bien connu est l’espèce Escherichia coli, qui comprend des souches commensales (et même probiotiques) ainsi que des souches très pathogènes (comme celles responsables du syndrome hémolytique et urémique [SHU]). En conséquence, pour déterminer et reconnaître les agents bactériens à haut risque de pathogénicité, et tracer les variants durant les évènements épidémiques, des solutions taxonomiques plus fines (au niveau des souches) sont nécessaires. Différents systèmes ont ainsi été utilisés pour distinguer les souches depuis les débuts de la microbiologie, comme les sérotypes, phagotypes, ribotypes ou autres ‘types’. Toutefois, avec la disponibilité récente des outils de séquençage génomique et la démocratisation de leur emploi, ces premiers systèmes tendent à disparaître au profit de nouvelles taxonomies à l’échelle des génomes.
Cet article présentera différents nouveaux systèmes de taxonomie génomique des souches bactériennes. Dans un article précédent (https://www.sfm-microbiologie.org/2023/02/14/comparaison-des-sequences-genomiques-pour-la-surveillance-epidemiologique-et-linvestigation-depidemies-3-approches-complementaires/), trois stratégies de comparaison de souches et de caractérisation de populations microbiennes ont été présentées : les approches gène-par-gène (Multi-Locus Sequence Typing; MLST), les techniques s’articulant sur les polymorphismes nucléotidiques (Single Nucleotide Polymorphisms; SNPs), et les méthodes basées sur les k-mers (oligonucléotides de longueur fixe k). Même si les stratégies basées sur les SNPs ou les k-mers ont permis, dans certains cas particuliers, de développer des taxonomies de souches (Wong et al. 2016; Lees et al. 2019; Dyson and Holt 2021; Hawkey et al. 2021), le typage MLST (Maiden et al. 1998; Achtman et al. 2012) demeure, pour les souches bactériennes, le système le plus généralisable et le plus utilisé en pratique. Son adaptation au core genome (cgMLST) a permis d’étendre les avantages bien établis de l’approche MLST à l’échelle génomique (Jolley and Maiden, 2010; Maiden et al., 2013). Ainsi, le typage cgMLST fournit une précision accrue, permettant une discrimination des souches à des niveaux beaucoup plus fins, notamment entre les isolats d’une même souche épidémique. Le présent article présentera donc uniquement les classifications et les nomenclatures associées aux approches gène-par-gène.
Le « Sequence Type » et son extension au core-genome MLST
Le « Sequence Type » (ST, que l’on pourrait traduire par « séquençotype ») est un génotype défini par l’approche MLST, traditionnellement fondée sur les séquences internes de ℓ = 7 gènes conservés dans une espèce donnée. Pour chacun des ℓ gènes, un numéro (entier) distinct est attribué à chaque nouvelle séquence nucléotidique (allèle) observée parmi les isolats. Pour chaque isolat, la combinaison des ℓ numéros d’allèles définit son profil allélique. Le ST étant un identifiant numérique unique attribué à chaque profil allélique complet, la classification des souches s’effectue via leur regroupement par ST. Du fait de la reproductibilité et de la portabilité de ce système, les classifications MLST se sont imposées comme des standards en biologie des populations et en épidémiologie des bactéries pathogènes, (e.g. https://pubmlst.org, https://enterobase.warwick.ac.uk, https://bigsdb.pasteur.fr). La liste des ST d’une espèce donnée représente donc une véritable nomenclature qui a été largement adoptée par les communautés des chercheurs et des acteurs de la surveillance épidémiologique des maladies infectieuses (Enright and Spratt 1999; Feil 2004; Achtman et al. 2012; Maiden et al. 2013).
Avec la démocratisation du séquençage à haut-débit des génomes, l’approche MLST a été logiquement étendue à l’échelle du core genome (cgMLST), le core genome étant l’ensemble des gènes partagés par tous les membres d’une espèce ou d’un groupe d’espèces. De façon analogue à la classification par ST, chaque profil cgMLST distinct (souvent appelé cgST) peut être répertorié. Toutefois, la différence majeure par rapport à l’approche MLST est l’existence potentielle de données manquantes au sein des profils cgMLST, du fait d’événements de délétions de gène, mais aussi de problèmes de séquençage (par exemple, faible profondeur de couverture locale) et/ou d’assemblage des séquences génomiques (gènes interrompus à l’extrémité d’un contig). Ainsi, des cgSTs ne différant que par des identifiants alléliques manquants pouvant coexister dans une nomenclature basée sur l’approche cgMLST, un même isolat peut être associé à plusieurs cgSTs différents.
Le regroupement des profils alléliques selon leur ressemblance
Une des limitations du génotypage basé sur l’entièreté du profil allélique est que les relations phylogénétiques entre souches ne sont pas déductibles à partir de simples identifiants (cg)ST. Ainsi, chez Klebsiella pneumoniae, les souches classifiées ST23 et ST57 sont connues pour être phylogénétiquement très proches, mais les numéros de ces deux ST ne traduisent pas cette information de proximité évolutive. Pour pallier cet aspect « typologiste » du (cg)ST, les profils alléliques peuvent être regroupés selon leur ressemblance. Cette approche a été concrétisée pour les profils MLST avec la notion de Complexe Clonal (CC), défini par l’ensemble des STs différant par un seul allèle d’au moins un autre ST du même CC. Ces regroupements en CC se sont avérés particulièrement utiles pour des pathogènes peu recombinogènes tels que Listeria monocytogenes ou Staphylococcus aureus, car les groupes d’isolats ainsi inférés sont génétiquement homogènes et bien séparés les uns des autres (Turner and Feil 2007; Ragon et al. 2008). Cependant, chez d’autres espèces telle que K. pneumoniae, la classification en CC s’est avérée vaine voire trompeuse, car de nombreuses paires de STs ne diffèrent que par un seul allèle (sur sept), produisant un effet de chaînage regroupant de manière trompeuse la plupart des STs dans un même CC très hétérogène (Turner et al. 2007; Brisse et al. 2009).
En généralisant cette approche par ressemblance à n’importe quel nombre de différences alléliques entre profils, des groupements de souches peuvent être définis pour tout niveau de divergence. Ainsi, dans l’approche cgMLST, des groupes de profils, parfois appelés groupes clonaux (Clonal Groups; CGs), sont déterminés en étendant la définition du CC à un seuil prédéfini appliqué à une dissimilarité entre profils (souvent estimée par la proportion d’allèles non-manquants différents). Cette méthode de détermination des CGs est souvent nommée classification Single Linkage (Slink; cf. Note technique).
Note technique
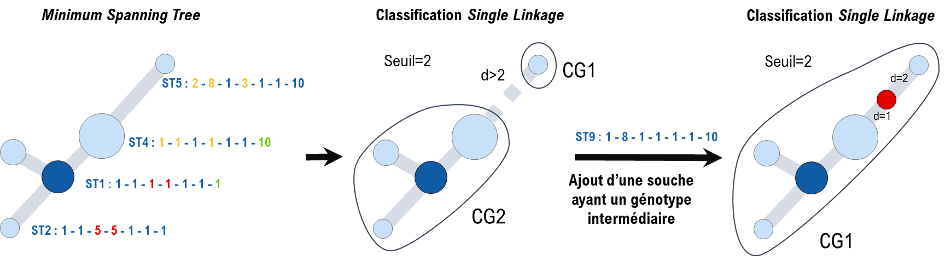
L’ensemble des dissimilarités entre toutes les paires de profils est équivalent à un graphe complet G, où chaque profil est représenté par un nœud et chaque arête (reliant deux nœuds distincts) a pour longueur la dissimilarité entre les deux profils associés. Cependant, le graphe G peut être remplacé par une représentation sous forme d’arbre. En effet, un arbre étant un graphe connexe (tous les sommets sont reliés) et sans cycle (chaque paire de sommet n’est reliée que par un unique chemin), cette représentation est généralement plus lisible qu’un graphe complet (beaucoup moins d’arêtes). Plus spécifiquement, G est souvent simplifié en utilisant un arbre couvrant de poids minimum (Minimum Spanning tree; MSTree; e.g. Graham and Hell, 1985), défini comme un arbre inclus dans G dont la somme des longueurs d’arête est minimale tout en reliant la totalité des nœuds de G. Ainsi, la représentation par MSTree (Figure 1) est très souvent privilégiée pour sa capacité à permettre de visualiser rapidement les proximités globales entre (cg)STs (Francisco et al. 2009). En complément, étant donné un seuil prédéfini, la suppression de toutes les arêtes du MSTree ayant une longueur supérieure à ce seuil conduit à la détermination de plusieurs composantes connexes (sous-graphes non-reliés) induisant chacune un regroupement de nœuds similaires (Figure 1, Zahn, 1971). De plus, ces regroupements de profils sont comparables à ceux induits par une classification hiérarchique par lien simple (Single Linkage; Slink; Gower and Ross, 1969).
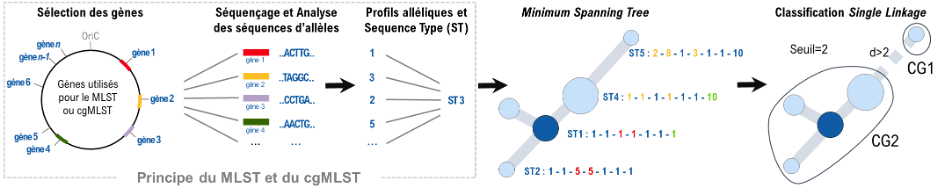
Figure 1. Principe du (cg)MLST, suivi de l’approche par arbre couvrant de poids minimum (MSTree) sur les dissimilarités entre profils alléliques. Notons que la taille de chaque nœud du MSTree est proportionnelle au nombre de profils (souches) associé(e)s au ST correspondant à ce nœud. Des groupes clonaux (CG) sont ensuite attribués à partir d’un seuil prédéfini (ici, seuil = 2). Ainsi, les groupes CG1 et CG2 représentent deux éléments d’une taxonomie.
Une limitation majeure de la classification Slink est son instabilité : l’apparition ultérieure d’un profil intermédiaire entre deux CGs préexistants implique la fusion des deux CGs (Figure 2). Des règles ad hoc peuvent être utilisées pour maintenir artificiellement la séparation des CGs destinés à fusionner. Cependant, ces règles partiellement arbitraires impliquent souvent une curation manuelle et non-reproductible, difficilement applicable aux jeux de données actuels de très grande taille qui dépassent le millier de profils distincts. Afin de pallier le problème d’instabilité de la classification Slink des profils cgMLST, trois stratégies différentes ont été proposées à ce jour.
Figure 2. Impact sur la classification Slink, de l’ajout d’une souche ayant un génotype intermédiaire et créant une fusion de groupes préexistants. Initialement, deux groupes CG1 et CG2 coexistent. Cependant, lors de l’apparition d’une souche ayant un génotype intermédiaire (représentée en rouge), cette souche présente une divergence inférieure ou égale au seuil avec un élément dans chacun des deux groupes CG1 et CG2. Suivant le paradigme de la classification Slink, les deux groupes doivent inclure ce dernier profil, impliquant leur fusion.
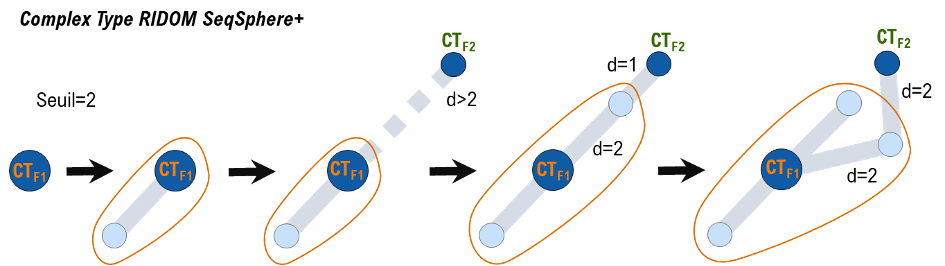
Le « Complex Type » (Ridom SeqSphere+)
Le Complex Type (CT) est un système de classification associé à Ridom SeqSphere+, un logiciel commercial dédié à l’analyse génomique microbienne, et relié aux nomenclatures cgMLST de nombreuses bactéries pathogènes (https://cgmlst.org/ncs). Un CT est défini par un seuil unique propre à chaque espèce bactérienne (par exemple, pour E. coli : 10 différences alléliques pour un schéma cgMLST de 2513 loci). En complément, chaque CT est déterminé par un profil nommé fondateur (static founder). Lorsque la dissimilarité entre un nouveau profil à classifier et un (unique) fondateur est inférieure ou égale au seuil préétabli, le nouveau profil est inclus dans le CT déterminé par ce fondateur ; sinon, ce nouveau profil devient le fondateur déterminant un nouveau CT (Figure 3). Toutefois, si, pour un nouveau profil à classifier, le critère d’inclusion est vérifié par plusieurs fondateurs en même temps, le CT avec le plus petit identifiant numérique sera privilégié (même si son fondateur n’est pas le plus proche du nouveau profil à classifier). Cette dernière règle de priorité peut malheureusement conduire à une fragmentation de la cohésion des regroupements entre isolats.
Figure 3. Classification par Complex Type (CT). La création des CTs suit le processus de soumission des profils cgMLST. Le premier fondateur CTF1 est créé avec le premier profil. Ensuite, si la distance entre un nouveau profil (bleu clair) et un fondateur (bleu foncé) est inférieure ou égale au seuil préétabli, ce nouveau profil est classifié dans le CT de ce fondateur. Sinon, un nouveau CT est établi et le nouveau profil devient le fondateur de ce nouveau CT (CTF2). A la dernière étape, le nouveau profil se trouvant à équidistance des deux fondateurs CTF1 et CTF2, il est affecté au CT avec le plus petit identifiant numérique (CTF1).
Les classifications stables multi-niveaux
La classification des souches à différents niveaux de profondeur phylogénétique est extrêmement utile, car elle permet d’examiner à la fois (i) les lignées profondes d’une espèce (par exemple, phylogroupes) pour comprendre leurs différences écologiques et leurs potentiels pathogènes (Wirth et al. 2006; Touchon et al. 2020), et (ii) les isolats très proches entre eux pour résoudre certaines questions épidémiologiques, telle que la détermination d’un lien entre des cas d’infections. Le développement récent de nouveaux systèmes de classification de souches permettant plusieurs niveaux de regroupement simultanés a ainsi pour but de répondre à ces besoins pluriels.
Quelques systèmes multi-niveaux ont déjà été proposés, telles que la méthode SNP address (Dallman et al. 2018), les approches à deux niveaux (Moura et al. 2016; Guglielmini et al. 2021) ou la technique Multilevel Single Linkage (MLSL, Figure 4, Hennart et al., 2022). Cependant, ces différentes propositions reposent toutes sur un principe de classification Slink, induisant donc des nomenclatures pouvant être instables (des regroupements peuvent fusionner au fur et à mesure de la découverte et de l’incorporation de nouveaux génomes, cf. Figure 2).
Figure 4. Nomenclature MLSL (MultiLevel Single Linkage). Dans cet exemple, deux seuils sont utilisés : seuilA=5 et seuilB=2. Pour chaque seuil, une décomposition du MStree est réalisée. Pour seuilA, tous les STs sont regroupés dans un seul groupe clonal CGA1. Pour seuilB, ST5 forme un groupe clonal distinct CGB1, alors que ST1, ST2, ST3 et ST4 forment un autre groupe clonal CGB2.
Deux méthodes cherchant explicitement à éviter le problème des fusions de groupes induites par l’introduction d’un génome intermédiaire ont ainsi été proposées : HierCC (Hierarchical Clustering of cgMLST; Zhou et al., 2021) et l’encodage LIN (Life Identification Numbers; Marakeby et al., 2014; Vinatzer et al., 2016, 2017; Tian et al., 2020; Hennart et al., 2022).
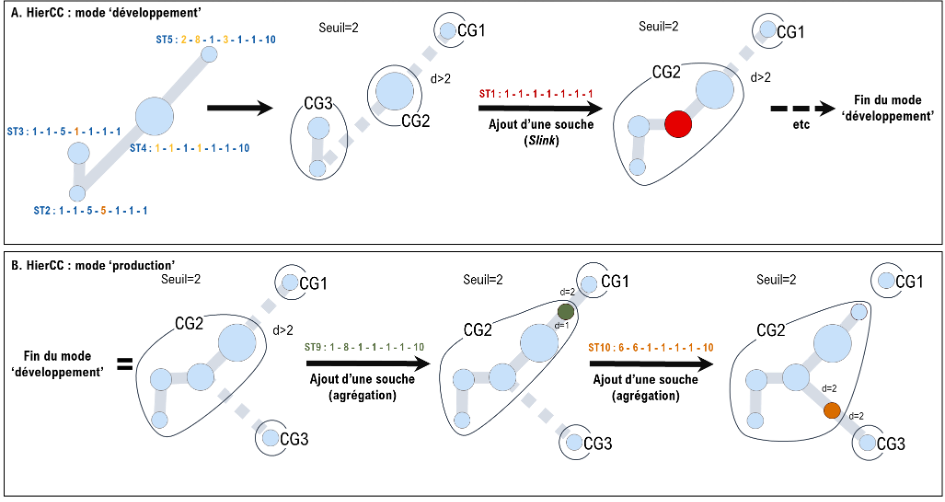
Nomenclature par clustering hiérarchique (HierCC)
L’approche HierCC peut être définie comme une classification Slink multi-niveaux, complétée par une stratégie pour compenser le problème de fusion des groupes. HierCC ajoute ainsi à la première étape de classification Slink (mode ‘développement’, autorisant les fusions de groupes), une seconde étape (mode ‘production’) où chaque nouveau profil est affecté au groupe préexistant le plus proche (contenant le profil le plus similaire au nouveau profil ; Figure 5). Dans ce dernier mode, si un nouveau profil est équidistant à plusieurs groupes préexistants, il est affecté au groupe ayant le plus petit identifiant numérique. En complément, HierCC propose une stratégie originale pour tenir compte des allèles manquants (Zhou, Charlesworth, and Achtman 2021).
Figure 5. Illustration de l’approche HierCC. (A) Mode ‘développement’ : les fusions de groupes sont autorisées. (B) Mode ‘production’ : aucune fusion n’est permise et chaque nouveau profil à classifier est affecté au groupe clonal (CG) contenant le profil déjà classifié le plus proche (panel B, étape 2). Si plusieurs CG sont équidistants, le CG le plus ancien (plus petit identifiant) est privilégié (panel B, étape 3).
Life Identification Numbers (LINs)
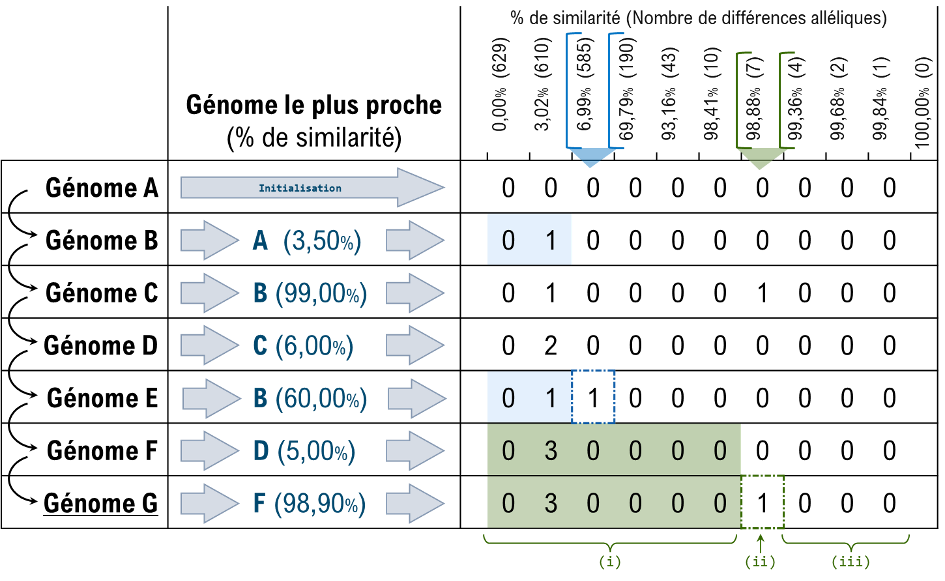
L’approche originelle de l’encodage LIN (LIN coding; Marakeby et al., 2014; Vinatzer et al., 2016, 2017; Tian et al., 2020) est basée sur une similarité globale entre paires de génomes assemblés (Average Nucleotide Identity; ANI; par exemple, Goris et al., 2007). Une adaptation basée sur les dissimilarités entre profils cgMLST (Figure 6) a ensuite été proposée (Hennart et al. 2022).
Figure 6. Procédure d’encodage LIN. Le système d’encodage LIN est défini par un ensemble de positions (niveaux) p, chacune correspondant à un seuil de similarité sp ∈ [0 , 1] tel que sp < sp+1. L’exemple de la figure reprend le système d‘encodage LIN à 10 positions dédié au schéma cgMLST de K. pneumoniae sensu lato (629 loci). Le code est tout d’abord initialisé (i.e. le premier génome est encodé par la valeur “0” à toutes les positions). Ensuite, la règle d’encodage d’un nouveau génome i à partir du plus proche j déjà encodé se définit à partir de la similarité sij ∈ [sp–1 , sp[ : (i) identique au code j jusqu’à la position p – 1 (incluse) ; (ii) pour la position p : valeur maximale observée à cette position (au sein du sous-ensemble des codes ayant le même préfixe à la position p – 1) incrémentée de 1 ; (iii) valeur “0” à partir de la position p + 1 (incluse) jusqu’à la dernière.
Dans le cadre de l’approche cgMLST, les LIN codes sont déterminés par un ensemble de positions (niveaux) définies par une fourchette de valeurs (croissantes) de similarités entre profils, allant de 0% (profils très distants, ne partageant aucun allèle commun) à 100% (profils identiques ; Figure 6). Chaque position est définie par un seuil inférieur (par exemple, 0% pour la première position) et est bornée par le seuil de la position suivante. Pour chaque espèce, le nombre de positions ainsi que les valeurs seuils définissant les différents niveaux doivent être déterminés en amont de tout codage. Cette définition initiale du système d’encodage LIN, propre à chaque espèce, peut être effectuée de manière arbitraire, mais des méthodes tenant compte de la structure de population des espèces considérées sont à privilégier. A cette fin, plusieurs outils ont été développés pour faciliter la détermination de seuils optimaux, tels que MSTclust (Hennart et al., 2022) ou HCCeval (Zhou et al., 2021).
Dans l’encodage LIN (comme dans HierCC), les préfixes (partie initiale en partant de la gauche) des codes multi-niveaux permettent d’identifier des groupes particuliers. Ces préfixes représentent donc des marqueurs diagnostics qui peuvent être utilisés pour identifier des lignées ou des clones d’importances médicale ou épidémiologique. Ainsi, des génomes très différents induisent des préfixes partagés très courts (voire aucun préfixe commun), alors que des génomes très similaires induisent des préfixes identiques jusqu’à une position proche de leur extrémité droite. Autrement dit, deux génomes sont d’autant plus similaires qu’ils présentent de plus longs préfixes identiques dans leurs codes multi-niveaux respectifs. Une proximité entre souches peut donc être déduite de la longueur de leur préfixe commun, deux isolats identiques ayant (par définition) exactement le même code.
Conclusion
La taxonomie des souches microbiennes est un domaine en évolution rapide. Les différentes approches taxonomiques existantes ont chacune leurs forces et leurs faiblesses, et le choix de la méthode appropriée dépend de divers facteurs, telle que la diversité génétique d’une espèce, mais aussi les outils de génotypage utilisés ou les plateformes génomiques disponibles. Les approches gène-par-gène (MLST ou cgMLST) demeurent très utiles pour la majorité des espèces bactériennes, génétiquement hétérogènes la plupart du temps. Toutefois, lorsque les souches considérées sont très peu diverses (pathogènes dits monomorphes tels que Yersinia pestis ou Salmonella enterica sérotype Typhi), ce type d’approches peut se révéler trop peu discriminant.
Dans ce contexte, les approches multi-niveaux connaissent actuellement un essor important car la taxonomie qu’elles induisent permet de répondre à un ensemble varié de questions, tels que les liens entre génotypes et phénotypes (virulence) ou le repérage de souches épidémiques plus ou moins anciennes. Ces nomenclatures multi-niveaux partagent l’idée originale de fournir une série d’identifiants de groupe (“codes-barres”) pour chaque souche, apportant ainsi des informations de regroupement à différents niveaux de profondeur phylogénétique. A l’avenir, comparer les deux approches multi-niveaux HierCC et LIN coding permettrait de déterminer le degré de compatibilité entre les classifications de souches qu’elles induisent. Leur adoption par la communauté internationale pour différentes espèces microbiennes demeurera néanmoins le test ultime qui décidera si ces stratégies représentent une solution pratique à la taxonomie génomique des souches.
Remerciements
Les auteurs remercient Federica Palma pour la coordination de la plateforme génomique BIGSdb-Pasteur (https://bigsdb.pasteur.fr), Brice Raffestin et Bryan Brancotte pour la maintenance de l’application informatique BIGSdb et des bases de données associées, Alexandra Moura, Virginie Passet et Valérie Bouchez pour la curation des bases de données MLST et cgMLST, et Sébastien Bridel pour ses contributions au déploiement du système LIN coding du complexe d’espèces Klebsiella pneumoniae.
Références bibliographiques
-
Achtman, Mark, John Wain, François-Xavier Weill, Satheesh Nair, Zhemin Zhou, Vartul Sangal, Mary G. Krauland, et al. 2012. “Multilocus Sequence Typing as a Replacement for Serotyping in Salmonella Enterica.” PLoS Pathog 8 (6): e1002776. https://doi.org/10.1371/journal.ppat.1002776.
-
Brisse, S., C. Fevre, V. Passet, S. Issenhuth-Jeanjean, R. Tournebize, L. Diancourt, and P. Grimont. 2009. “Virulent Clones of Klebsiella Pneumoniae: Identification and Evolutionary Scenario Based on Genomic and Phenotypic Characterization.” PLoS One 4 (3): e4982. https://doi.org/10.1371/journal.pone.0004982.
-
Dallman, Timothy, Philip Ashton, Ulf Schafer, Aleksey Jironkin, Anais Painset, Sharif Shaaban, Hassan Hartman, et al. 2018. “SnapperDB: A Database Solution for Routine Sequencing Analysis of Bacterial Isolates.” Bioinformatics (Oxford, England) 34 (17): 3028–29. https://doi.org/10.1093/bioinformatics/bty212.
-
Dyson, Zoe A, and Kathryn E Holt. 2021. “Five Years of GenoTyphi: Updates to the Global Salmonella Typhi Genotyping Framework.” The Journal of Infectious Diseases 224 (Supplement_7): S775–80. https://doi.org/10.1093/infdis/jiab414.
-
Enright, M. C., and B. G. Spratt. 1999. “Multilocus Sequence Typing.” Trends Microbiol 7 (12): 482–87.
-
Feil, E. J. 2004. “Small Change: Keeping Pace with Microevolution.” Nat. Rev. Microbiol. 2 (6): 483–95.
-
Francisco, Alexandre P, Miguel Bugalho, Mário Ramirez, and João A Carriço. 2009. “Global Optimal EBURST Analysis of Multilocus Typing Data Using a Graphic Matroid Approach.” BMC Bioinformatics 10 (1): 152. https://doi.org/10.1186/1471-2105-10-152.
-
Goris, Johan, Konstantinos T. Konstantinidis, Joel A. Klappenbach, Tom Coenye, Peter Vandamme, and James M. Tiedje. 2007. “DNA–DNA Hybridization Values and Their Relationship to Whole-Genome Sequence Similarities.” International Journal of Systematic and Evolutionary Microbiology, 57 (1): 81–91. https://doi.org/10.1099/ijs.0.64483-0.
-
Gower, J. C., and G. J. S. Ross. 1969. “Minimum Spanning Trees and Single Linkage Cluster Analysis.” Applied Statistics 18 (1): 54. https://doi.org/10.2307/2346439.
-
Graham, R.L., and Pavol Hell. 1985. “On the History of the Minimum Spanning Tree Problem.” IEEE Annals of the History of Computing 7 (1): 43–57. https://doi.org/10.1109/MAHC.1985.10011.
-
Guglielmini, Julien, Melanie Hennart, Edgar Badell, Julie Toubiana, Alexis Criscuolo, and Sylvain Brisse. 2021. “Genomic Epidemiology and Strain Taxonomy of Corynebacterium Diphtheriae.” Journal of Clinical Microbiology 59 (12): e0158121. https://doi.org/10.1128/JCM.01581-21.
-
Hawkey, Jane, Kalani Paranagama, Kate S. Baker, Rebecca J. Bengtsson, François-Xavier Weill, Nicholas R. Thomson, Stephen Baker, et al. 2021. “Global Population Structure and Genotyping Framework for Genomic Surveillance of the Major Dysentery Pathogen, Shigella Sonnei.” Nature Communications 12 (1): 2684. https://doi.org/10.1038/s41467-021-22700-4.
-
Hennart, Melanie, Julien Guglielmini, Sébastien Bridel, Martin C J Maiden, Keith A. Jolley, Alexis Criscuolo, and Sylvain Brisse. 2022. “A Dual Barcoding Approach to Bacterial Strain Nomenclature: Genomic Taxonomy of Klebsiella Pneumoniae Strains.” Molecular Biology and Evolution 39 (7): msac135. https://doi.org/10.1093/molbev/msac135.
-
Jolley, Keith A., and Martin CJ Maiden. 2010. “BIGSdb: Scalable Analysis of Bacterial Genome Variation at the Population Level.” BMC Bioinformatics 11 (1): 595. https://doi.org/10.1186/1471-2105-11-595.
-
Konstantinidis, Konstantinos T., Alban Ramette, and James M. Tiedje. 2006. “Toward a More Robust Assessment of Intraspecies Diversity, Using Fewer Genetic Markers.” Applied and Environmental Microbiology 72 (11): 7286–93. https://doi.org/10.1128/AEM.01398-06.
-
Lees, John A., Simon R. Harris, Gerry Tonkin-Hill, Rebecca A. Gladstone, Stephanie W. Lo, Jeffrey N. Weiser, Jukka Corander, Stephen D. Bentley, and Nicholas J. Croucher. 2019. “Fast and Flexible Bacterial Genomic Epidemiology with PopPUNK.” Genome Research 29 (2): 304–16. https://doi.org/10.1101/gr.241455.118.
-
Maiden, M. C., J. A. Bygraves, E. Feil, G. Morelli, J. E. Russell, R. Urwin, Q. Zhang, et al. 1998. “Multilocus Sequence Typing: A Portable Approach to the Identification of Clones within Populations of Pathogenic Microorganisms.” Proc. Natl. Acad. Sci. U. S. A. 95 (6): 3140–45.
-
Maiden, M. C., M. J. van Rensburg, J. E. Bray, S. G. Earle, S. A. Ford, K. A. Jolley, and N. D. McCarthy. 2013. “MLST Revisited: The Gene-by-Gene Approach to Bacterial Genomics.” Nat Rev Microbiol 11 (10): 728–36. https://doi.org/10.1038/nrmicro3093.
-
Marakeby, Haitham, Eman Badr, Hanaa Torkey, Yuhyun Song, Scotland Leman, Caroline L. Monteil, Lenwood S. Heath, and Boris A. Vinatzer. 2014. “A System to Automatically Classify and Name Any Individual Genome-Sequenced Organism Independently of Current Biological Classification and Nomenclature.” PloS One 9 (2): e89142. https://doi.org/10.1371/journal.pone.0089142.
-
Moura, Alexandra, Alexis Criscuolo, Hannes Pouseele, Mylène M. Maury, Alexandre Leclercq, Cheryl Tarr, Jonas T. Björkman, et al. 2016. “Whole Genome-Based Population Biology and Epidemiological Surveillance of Listeria Monocytogenes.” Nature Microbiology 2 (October): 16185. https://doi.org/10.1038/nmicrobiol.2016.185.
-
Payne, Michael, Sandeep Kaur, Qinning Wang, Daneeta Hennessy, Lijuan Luo, Sophie Octavia, Mark M. Tanaka, Vitali Sintchenko, and Ruiting Lan. 2020. “Multilevel Genome Typing: Genomics-Guided Scalable Resolution Typing of Microbial Pathogens.” Eurosurveillance 25 (20): 1900519. https://doi.org/10.2807/1560-7917.ES.2020.25.20.1900519.
-
Ragon, M., T. Wirth, F. Hollandt, R. Lavenir, M. Lecuit, A. Le Monnier, and S. Brisse. 2008. “A New Perspective on Listeria Monocytogenes Evolution.” PLoS Pathog 4 (9): e1000146.
-
Tian, Long, Chengjie Huang, Reza Mazloom, Lenwood S. Heath, and Boris A. Vinatzer. 2020. “LINbase: A Web Server for Genome-Based Identification of Prokaryotes as Members of Crowdsourced Taxa.” Nucleic Acids Research 48 (W1): W529–37. https://doi.org/10.1093/nar/gkaa190.
-
Touchon, Marie, Amandine Perrin, Jorge André Moura De Sousa, Belinda Vangchhia, Samantha Burn, Claire L. O’Brien, Erick Denamur, David Gordon, and Eduardo Pc Rocha. 2020. “Phylogenetic Background and Habitat Drive the Genetic Diversification of Escherichia Coli.” Edited by Xavier Didelot. PLOS Genetics 16 (6): e1008866. https://doi.org/10.1371/journal.pgen.1008866.
-
Turner, K. M., and E. J. Feil. 2007. “The Secret Life of the Multilocus Sequence Type.” Int J Antimicrob Agents 29 (2): 129–35.
-
Turner, K. M., W. P. Hanage, C. Fraser, T. R. Connor, and B. G. Spratt. 2007. “Assessing the Reliability of EBURST Using Simulated Populations with Known Ancestry.” BMC Microbiol 7: 30.
-
Vinatzer, Boris A., Long Tian, and Lenwood S. Heath. 2017. “A Proposal for a Portal to Make Earth’s Microbial Diversity Easily Accessible and Searchable.” Antonie van Leeuwenhoek 110 (10): 1271–79. https://doi.org/10.1007/s10482-017-0849-z.
-
Vinatzer, Boris A., Alexandra J. Weisberg, Caroline L. Monteil, Haitham A. Elmarakeby, Samuel K. Sheppard, and Lenwood S. Heath. 2016. “A Proposal for a Genome Similarity-Based Taxonomy for Plant-Pathogenic Bacteria That Is Sufficiently Precise to Reflect Phylogeny, Host Range, and Outbreak Affiliation Applied to Pseudomonas Syringae Sensu Lato as a Proof of Concept.” Phytopathology 107 (1): 18–28. https://doi.org/10.1094/PHYTO-07-16-0252-R.
-
Wirth, Thierry, Daniel Falush, Ruiting Lan, Frances Colles, Patience Mensa, Lothar H. Wieler, Helge Karch, et al. 2006. “Sex and Virulence in Escherichia Coli: An Evolutionary Perspective.” Molecular Microbiology 60 (5): 1136–51. https://doi.org/10.1111/j.1365-2958.2006.05172.x.
-
Wong, Vanessa K., Stephen Baker, Thomas R. Connor, Derek Pickard, Andrew J. Page, Jayshree Dave, Niamh Murphy, et al. 2016. “An Extended Genotyping Framework for Salmonella Enterica Serovar Typhi, the Cause of Human Typhoid.” Nature Communications 7 (1): 12827. https://doi.org/10.1038/ncomms12827.
-
Zahn, C.T. 1971. “Graph-Theoretical Methods for Detecting and Describing Gestalt Clusters.” IEEE Transactions on Computers C–20 (1): 68–86. https://doi.org/10.1109/T-C.1971.223083.
-
Zhou, Zhemin, Jane Charlesworth, and Mark Achtman. 2021. “HierCC: A Multi-Level Clustering Scheme for Population Assignments Based on Core Genome MLST.” Bioinformatics (Oxford, England), April, btab234. https://doi.org/10.1093/bioinformatics/btab234.