La saga de l’OXA-48 : voyage express chez les entérobactéries
François Gravey,1,2 et Sylvain Brisse3
1. Université Caen Normandie, Dynamicure, INSERM UMR U1311, Caen, France
2. CHU de Caen Normandie, Service de bactériologie, Caen, France
3. Institut Pasteur, Université Paris Cité, Unité de recherche Biodiversité et Épidémiologie des Bactéries Pathogènes, Paris, France
Section épidémiologie et génomique des populations de la SFM
L’enzyme oxacillinase 48 (OXA-48) appartient à la classe D des beta-lactamases selon la classification d’Ambler (1). Elle a pour origine l’OXA-54 présente dans le chromosome de Shewanella oneidensis (2). L’OXA-48 a été décrite pour la première fois en 2001 sur un plasmide porté par une souche de Klebsiella pneumoniae isolée à partir d’un examen cytobactériologique des urines à Istanbul en Turquie (3). Responsable d’épisodes sporadiques puis épidémiques en Turquie, elle a été retrouvée à partir des années 2010 au sein de très nombreuses espèces d’Entérobactéries : Escherichia coli, Enterobacter cloacae complex, Providencia rettgeri, Citrobacter freundii et sur tous les continents (4). Dans les pays du bassin méditerranéen, la circulation de cette carbapénèmase est considérée comme endémique, et est responsable d’épidémies intra-hospitalières en Europe, en Afrique du Sud, en Chine et en Australie (4). L’OXA-48 et ses variants (cf. infra) sont les carbapénèmases les plus fréquemment retrouvées en France. Dans une étude réalisée en 2014, les enzymes de type OXA-48-like représentaient 86% d’un panel de plus de 1000 souches françaises d’entérobactéries productrices de carbapénèmases (EPC), très loin devant les New-Delhi Metallo-beta-lactamases NDM (8%) et les Klebsiella pneumoniae carbapenemase (KPC) (2%) (5). Le groupe European Survey of Carbapenemase-Producing Enterobacteriaceae (EuSCAPE) classait la situation française au stade quatre, sur une échelle qui en compte cinq, vis-à-vis de la dissémination des OXA-48, c’est-à-dire la présence d’une dissémination interrégionale de l’enzyme à l’échelle du territoire national (6).
L’OXA-48 a une activité hydrolytique vis-à-vis des pénicillines et des carbapénèmes dont l’imipénème, l’ertapénème et le méropénème mais peu ou pas d’activité vis-à-vis des céphalosporines de troisième génération (7). En l’absence d’autre mécanisme de résistance associé, la détection des souches productrices d’OXA-48 peut être difficile du fait de : (i) la faible hydrolyse des carbapénèmes qui peut alors passer inaperçue, et (ii) l’absence de tests phénotypiques simples tels que ceux basés sur les inhibiteurs de beta-lactamases (8). C’est lorsque l’OXA-48 est associée avec d’autres mécanismes de résistances tels que la production de béta-lactamases à spectre étendu (BLSE), une hyperproduction de la céphalosporinase AmpC, une hyperproduction de pompes d’efflux et/ou la fermeture ou perte de porines que les problématiques thérapeutiques se posent (9). Une étude des souches productrices de l’OXA-48 dans les pays Européens et Nord-Africains retrouvait une co-production de l’OXA-48 avec une BLSE dans 75% des cas, principalement la CTX-M-15 (10). Retrouvés chez différentes espèces d’entérobactéries et fréquemment associées à d’autres mécanismes de résistance, les antibiogrammes des souches productrices de l’OXA-48 peuvent être très variables. Il est néanmoins important de noter l’hydrolyse de la témocilline (Figure 1).
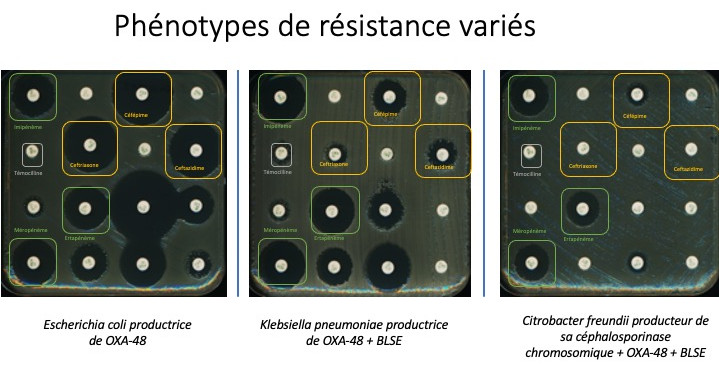
Fréquemment isolées à partir de prélèvements de dépistage tels que les écouvillonnages rectaux, les souches productrices d’OXA-48 peuvent par la suite être responsables d’infections profondes dans des proportions variables selon les travaux, de 18 à 65% (7, 11). Une étude réalisée aux Pays-Bas retrouvait un taux de mortalité toutes causes confondues de 30% parmi les patients infectés par une souche productrice d’OXA-48 (7).
Actuellement, la base de données des beta-lactamases http://bldb.eu/ contient plus de 50 enzymes décrites comme variants de l’OXA-48, qui constituent la famille des OXA-48 like (12). Ces enzymes présentent des capacités d’hydrolyse pouvant être différentes et certaines ne sont même plus des carbapénèmases mais présentent une affinité uniquement vis-à-vis des C3G, par exemple : OXA-163 et OXA-405 (12, 13).
Les données de la littérature à propos d’épisodes épidémiques de souches productrices d’OXA-48 retrouvent très fréquemment un rapport élevé entre le nombre d’espèces bactériennes isolées et le nombre de patients. Selon les travaux, de 22% à 47% des patients portaient au moment du diagnostic d’EPC plusieurs espèces productrices de OXA-48 allant parfois jusqu’à cinq de façon concomitante (7, 8). A l’inverse des autres types d’enzymes, telles que les NDM, KPC, Verona IMipenase (VIM), dont la dissémination est fréquemment associée à un clone bactérien épidémique, la diffusion de l’OXA-48 a une dynamique populationnelle différente, avant tout basée sur la diffusion du gène de l’enzyme à travers de nombreuses espèces au sein d’un hôte. Cette particularité explique les très nombreuses populations d’entérobactéries au sein desquelles l’OXA-48 a été retrouvée (14).
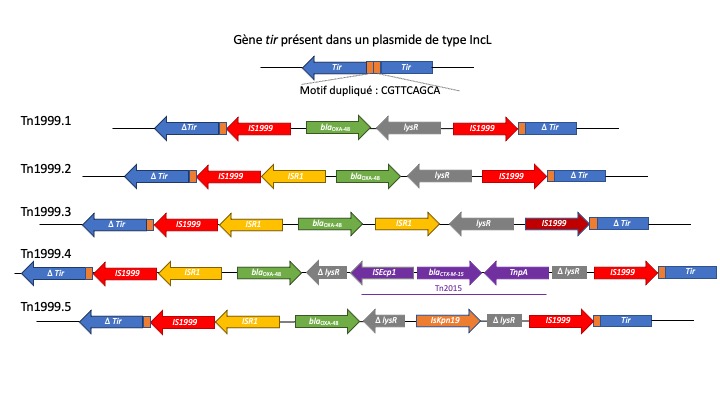
Ces données s’expliquent par les supports génomiques associés à l’OXA-48. Le gène de l’OXA-48 est inséré au sein du transposon Tn1999 dont il existe actuellement cinq formes variantes (Figure 2) (4). Dans sa forme la plus simple Tn1999.1, le transposon est composé de deux séquences d’insertion IS1999 en miroir qui entourent le gène blaOXA-48, un gène codant pour un régulateur transcriptionnel LysR, et un fragment d’un gène codant pour une acetyl coenzyme A carboxylase. Les transposons Tn1999.2 et Tn1999.3 portent en plus, tous les deux, une copie de la séquence d’insertion IS1R en amont du gène blaOXA-48 (Figure 2). Le Tn1999.3 aussi porte une seconde copie de l’IS1R située entre les gènes blaOXA-48 et lysR. La présence de la séquence d’insertion IS1R en amont du gène blaOXA-48 créé un promoteur fort responsable d’une activité enzymatique deux fois plus importante (Figure 2) (4). Les transposons Tn1999.4 et Tn1999.5 sont tous les deux des variants du Tn1999.2. Dans chacun d’entre eux, le gène lysR est tronqué par l’insertion du transposon Tn2015 porteur du gène de la BLSE blaCTX-M-15 (variant Tn1999.4) et la séquence d’insertion ISKpn19 (Tn1999.5) (Figure 2). Dans une étude portant sur plus de 100 souches productrices de OXA-48 isolées entre 2001 et 2011 en Europe et en Afrique du Nord, le Transposon Tn1999.2 était le variant le plus fréquemment retrouvé (78%) (10). Le motif génomique d’insertion reconnu par le Tn1999 consiste en une duplication d’un motif cible de neuf paires de bases : CGTTCAGCA présentes dans le gène tir, qui code pour protéine d’inhibition de transfert (15, 16). Des études de conjugaison ont démontré que l’interruption du gène tir par l’insertion du transposon Tn1999 engendre la synthèse d’une protéine non fonctionnelle et de ce fait, une augmentation de 50 à 100 fois le taux de transfert conjugatif du plasmide (16).
Les transposons Tn1999 porteurs de l’OXA-48 sont retrouvés au sein de plasmides de type IncL dans plus de 90% des cas tandis que les autres carbapénèmases telles que KPC et NDM sont souvent trouvées sur des plasmides appartenant aux groupes IncF et IncN (10, 17). Les plasmides IncL mesurent entre 60 et 70 kb et, classiquement, ils ne contiennent pas d’autres gènes de résistance aux antibiotiques et sont en faible nombre de copies dans la cellule (8, 15, 17, 18). Ils portent également des opérons de transfert tra et trb qui codent pour les pili de type IV impliqués dans le mécanisme de conjugaison. Des travaux ont démontré le possible transfert de ces plasmides entre espèces d’Entérobactéries mais n’ont pas réussi à transmettre le plasmide à des souches de Pseudomonas aeruginosa et Acinetobacter baumannii. Ces données peuvent expliquer l’association de l’OXA-48 avec les entérobactéries (15). Certains de ces plasmides sont bien décrits dans la littérature : E71T isolé lors d’une épidémie de K. pneumoniae productrices de OXA-48 en Irlande ou pOXA-48-4963A dans une étude réalisée en république Tchèque (8, 19). Les OXA-48 ont été retrouvées dans d’autres types de plasmides que IncL, chez lesquels l’insertion du transposon ne s’est pas effectuée sur le gène tir. Par exemple, le transposon Tn1999 a été retrouvé au sein d’un plasmide non groupable de 150 kb isolé d’une souche de K. pneumoniae, dans lequel Tn1999 interrompait un gène codant pour une phosphoadenosine phosphosulfate reductase (8, 18).
Enfin, des intégrations chromosomiques de l’OXA-48 ont été décrites. L’insertion de l’enzyme se fait via le transposon Tn6237, notamment au sein du clone de Escherichia coli ST38 (20).
Les souches productrices d’OXA-48 n’ont pas été exclusivement isolées de prélèvements humains. En effet, des études ont retrouvé des bactéries productrices d’OXA-48 à partir de prélèvements environnementaux réalisés dans les hôpitaux en contexte épidémique : surfaces, matelas, éviers, toilettes ; mais aussi à partir d’eaux usées, ou d’animaux de compagnie (chats, chiens, chevaux) (14). Tous ces réservoirs constituent des routes de transmission possibles jusqu’à l’humain, mais sont sans doute plutôt des contaminants secondaires à partir d’humains.
Conclusions :
Le diagnostic de laboratoire de l’OXA-48 n’est pas toujours évident du fait de sa faible hydrolyse des carbapénèmes et l’absence de tests phénotypiques dédiés. Sur les plasmides de type IncL, le transposon Tn1999 s’insère au sein du gène tir, ce qui a pour effet de décupler les capacités conjugatives des plasmides et explique l’ampleur de la diffusion de l’OXA-48. Autres conséquences, les patients sont souvent porteurs au même moment de différentes espèces productrices d’OXA-48. Tous ces éléments concourent à une grande capacité épidémique de ce mécanisme de résistance. Aussi, la présence de plusieurs espèces productrices d’OXA-48 n’est que faussement rassurante et doit déclencher des explorations plus précises sur les liens épidémiologiques entre souches et plasmides, s’appuyant sur de nouvelles technologies de séquençage à haut débit, y compris de troisième génération (‘long read sequencing’).
Bibliographie
-
Ramette A, Gasser M, Nordmann P, Zbinden R, Schrenzel J, Perisa D, Kronenberg A. 2021. Temporal and regional incidence of carbapenemase-producing Enterobacterales, Switzerland, 2013 to 2018. Eurosurveillance 26.
-
Poirel L, Héritier C, Nordmann P. 2004. Chromosome-Encoded Ambler Class D β-Lactamase of Shewanella oneidensis as a Progenitor of Carbapenem-Hydrolyzing Oxacillinase. Antimicrob Agents Chemother 48:348–351.
-
Poirel L, Héritier C, Tolün V, Nordmann P. 2004. Emergence of Oxacillinase-Mediated Resistance to Imipenem in Klebsiella pneumoniae. Antimicrob Agents Chemother 48:15–22.
-
Pitout JDD, Peirano G, Kock MM, Strydom K-A, Matsumura Y. 2019. The Global Ascendency of OXA-48-Type Carbapenemases. Clin Microbiol Rev 33:e00102-19.
-
Dortet L, Cuzon G, Ponties V, Nordmann P. 2017. Trends in carbapenemase-producing Enterobacteriaceae, France, 2012 to 2014. Eurosurveillance 22.
-
Albiger B, Glasner C, Struelens MJ, Grundmann H, Monnet DL, the European Survey of Carbapenemase-Producing Enterobacteriaceae (EuSCAPE) working group. 2015. Carbapenemase-producing Enterobacteriaceae in Europe: assessment by national experts from 38 countries, May 2015. Eurosurveillance 20.
-
Dautzenberg MJ, Ossewaarde JM, de Kraker ME, van der Zee A, van Burgh S, de Greeff SC, Bijlmer HA, Grundmann H, Cohen Stuart JW, Fluit AC, Troelstra A, Bonten M. 2014. Successful control of a hospital-wide outbreak of OXA-48 producing Enterobacteriaceae in the Netherlands, 2009 to 2011. Eurosurveillance 19.
-
Skalova A, Chudejova K, Rotova V, Medvecky M, Studentova V, Chudackova E, Lavicka P, Bergerova T, Jakubu V, Zemlickova H, Papagiannitsis CC, Hrabak J. 2017. Molecular Characterization of OXA-48-Like-Producing Enterobacteriaceae in the Czech Republic and Evidence for Horizontal Transfer of pOXA-48-Like Plasmids. Antimicrob Agents Chemother 61:e01889-16.
-
Nordmann P, Naas T, Poirel L. 2011. Global Spread of Carbapenemase-producing Enterobacteriaceae. Emerg Infect Dis 17:1791–1798.
-
Potron A, Poirel L, Rondinaud E, Nordmann P. 2013. Intercontinental spread of OXA-48 beta-lactamase-producing Enterobacteriaceae over a 11-year period, 2001 to 2011. Eurosurveillance 18.
-
Jolivet S, Couturier J, Vuillemin X, Gouot C, Nesa D, Adam M, Brissot E, Mohty M, Bonnin RA, Dortet L, Barbut F. 2021. Outbreak of OXA-48-producing Enterobacterales in a haematological ward associated with an uncommon environmental reservoir, France, 2016 to 2019. Eurosurveillance 26.
-
Dabos L, Oueslati S, Bernabeu S, Bonnin RA, Dortet L, Naas T. 2022. To Be or Not to Be an OXA-48 Carbapenemase. Microorganisms 10:258.
-
Oueslati S, Retailleau P, Marchini L, Dortet L, Bonnin RA, Iorga BI, Naas T. 2019. Biochemical and Structural Characterization of OXA-405, an OXA-48 Variant with Extended-Spectrum β-Lactamase Activity. Microorganisms 8:24.
-
Mairi A, Pantel A, Sotto A, Lavigne J-P, Touati A. 2018. OXA-48-like carbapenemases producing Enterobacteriaceae in different niches. Eur J Clin Microbiol Infect Dis 37:587–604.
-
Poirel L, Bonnin RA, Nordmann P. 2012. Genetic Features of the Widespread Plasmid Coding for the Carbapenemase OXA-48. Antimicrob Agents Chemother 56:559–562.
-
Potron A, Poirel L, Nordmann P. 2014. Derepressed Transfer Properties Leading to the Efficient Spread of the Plasmid Encoding Carbapenemase OXA-48. Antimicrob Agents Chemother 58:467–471.
-
Kopotsa K, Osei Sekyere J, Mbelle NM. 2019. Plasmid evolution in carbapenemase‐producing Enterobacteriaceae : a review. Ann N Y Acad Sci 1457:61–91.
-
Carrër A, Poirel L, Yilmaz M, Akan ÖA, Feriha C, Cuzon G, Matar G, Honderlick P, Nordmann P. 2010. Spread of OXA-48-Encoding Plasmid in Turkey and Beyond. Antimicrob Agents Chemother 54:1369–1373.
-
Power K, Wang J, Karczmarczyk M, Crowley B, Cotter M, Haughton P, Lynch M, Schaffer K, Fanning S. 2014. Molecular Analysis of OXA-48-Carrying Conjugative IncL/M-Like Plasmids in Clinical Isolates of Klebsiella pneumoniae in Ireland. Microb Drug Resist 20:270–274.
-
Turton JF, Doumith M, Hopkins KL, Perry C, Meunier D, Woodford N. 2016. Clonal expansion of Escherichia coli ST38 carrying a chromosomally integrated OXA-48 carbapenemase gene. J Med Microbiol 65:538–546.








